基因组jpboy
jpboy 时间:2021-01-26 阅读:()
HEREDITAS(Beijing)2011年8月,33(8):847―856ISSN0253-9772www.
chinagene.
cn综述收稿日期:20000000;修回日期:20000000基金项目:国家高技术研究发展计划项目(863计划)(编号:2007AA02Z161)资助作者简介:张鑫,博士,研究方向:复杂疾病遗传学.
E-mail:zhangxin68619@163.
com通讯作者:张学军,博士,主任医师,教授,博士生导师,研究方向:复杂疾病遗传学.
E-mail:ayzxj@vip.
sina.
comDOI:10.
3724/SP.
J.
1005.
2011.
00847全基因组外显子测序及其应用张鑫,李敏,张学军安徽医科大学皮肤病研究所,合肥230032摘要:近年来,众多研究小组开展了大量的全基因组关联研究(Genome-wideassociationstudies,GWAS),发现并鉴定了许多与复杂疾病/性状相关联的遗传变异,为复杂疾病发病机制的研究提供了重要线索.
由于GWAS的结果存在假阳性、假阴性、检测到的单核苷酸多态性很少位于功能区以及对稀有变异和结构变异不敏感等问题,导致了其应用的局限性.
而新一代测序技术的进步,促进了全基因组测序和全基因组外显子测序的快速发展,为解决上述问题提供了契机.
全基因组外显子测序是利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法.
由于其具有对常见和罕见变异高灵敏度,能发现外显子区绝大部分疾病相关变异以及仅需要对约1%的基因组进行测序等优点,促使全基因组外显子测序成为鉴定孟德尔疾病的致病基因最有效的策略,也被运用于复杂疾病易感基因的研究和临床诊断中.
关键词:外显子测序;孟德尔疾病;复杂疾病;临床诊断ExomesequencinganditsapplicationZHANGXin,LIMin,ZHANGXue-JunInstituteofDermatologyofAnhuiMedicalUniversity,Hefei230032,ChinaAbstract:Inrecentyears,researchershaveidentifiedalargenumberofcomplexdiseases/traits-associatedgeneticvari-antsbyperforminggenome-wideassociationstudies(GWAS),whichmayprovideimportantcluesonunderstandingthemechanismsofrelateddiseases.
However,GWAShasitsownlimitationsintermsofbeingfalsepositive,falsenegativeresults,veryfewSNPslocatedinthefunctionalareasandinsensitivetodetectrareandstructuralvariations,whichresultsintheapplicationlimitationofthismethod.
Withthedevelopmentofthenext-generationsequencingtechnology,wholege-nomeandexomesequencingdevelopedrapidlyandprovideanopportunityforustodealwiththeproblemcausedbyGWAS.
Thishigh-throughputsequencingtechnologyisappliedforsequencingtheexome(1%ofgenome)todiscovermostofthediseases-relatedvariationsinexons.
Furthermore,itishighlyeffectivetodetectcommonandrarevariations.
Duetotheseadvantages,exomesequencinghasbecomeapowerfulandefficientstrategyforidentifyingthegenesresponsibleformendeliandisordersandcomplexdiseases,whichwillbeveryhelpfulforthediseasesclinicaldiagnosis.
Keywords:exomesequencing;mendeliandisorders;complexdiseases;clinicaldiagnosis随着人类基因组计划(Humangenomeproject,HGP)和国际人类基因组单体型图计划(Theinterna-tionalHapMapproject)的完成以及高通量生物芯片技术的成功研发,人们广泛利用高通量全基因组生848HEREDITAS(Beijing)2011第33卷物芯片的技术手段,采用关联分析的方法即全基因组关联研究(Genome-wideassociationstudies,GWAS)来筛选复杂疾病易感基因,取得了前所未有的成就.
但由于GWAS存在着一定的局限性,同时随着新一代测序技术的迅猛发展,促进了全基因组测序和全基因组外显子测序的发展.
本文就全基因组外显子测序的研究方法及其应用作一综述.
1外显子测序的产生背景GWAS是通过对大规模的群体(病例/对照)DNA样本进行包括单核苷酸多态性变异(Single-nucleotidepolymorphisms,SNPs)、拷贝数变异(Copynumbervariation,CNV)在内的全基因组高密度遗传标记并分型,从而寻找与复杂疾病相关的遗传因素的研究方法,掀起了人类基因组研究第3次浪潮.
自2005年以来,利用GWAS对多种常见疾病进行了研究,发现和重复验证了近2000个SNPs或位点,其中包括以前未检测到的而与疾病密切相关的基因及部分未知基因[1].
但此方法有其欠缺,如容易产生假阳性和假阴性,而且发现的与疾病关联的SNPs多位于基因间或内含子上,很少位于功能区(如外显子区和5'UTR区).
同时,芯片检测位点有一定的有限性,除Illumina的新一代全基因组基因分型芯片外,多数是发现常见变异(Minorallelefrequency,MAF>5%),而对稀有变异(MAF<5%)和其他结构变异不敏感.
目前,GWAS是基于常见疾病/常见变异的假说,而越来越多的研究结果表明许多复杂疾病是由稀有变异造成的[2,3],而这种基于芯片的GWAS在实验设计时尚未充分考虑这部分信息,故而较难搜寻稀有变异.
过去主要通过定位克隆、物理作图和候选基因测序的方法研究孟德尔疾病,但这些方法受到诸多因素的限制,如患者人数少、家系少或小、有意识的减少生育等,许多孟德尔疾病尚未发现其致病基因.
现有的技术和研究方法在研究孟德尔疾病和复杂疾病时均遇见了难以解决的问题,迫切需要新技术的出现.
而随着新一代测序技术的迅速发展、测序费用降低和时间缩短,使得全基因组和全基因组外显子测序在大规模人群中的运用成为现实.
由于全基因组测序的费用在短时间内很难降到与外显子测序相当的价格,在经费一定的情况下,全基因组外显子测序更适合大批量样本研究以获得高深度的测序数据.
更重要的是,目前一致认为大部分功能变异都潜藏在外显子中[4],这是因为引起孟德尔疾病的突变主要位于基因内造成的[4,5].
因此,全基因组外显子测序已成为现阶段基因测序工作的重心.
2外显子测序的基本思路及技术路线外显子测序主要包括目标区域序列的富集、DNA测序、生物信息学统计3个主要步骤.
2.
1目标区域序列的富集在过去的20年里,传统的Sanger测序主要是利用UniplexPCR和MutliplexPCR方法富集目标区域序列[6],但其引物合成与PCR反应所需费用都很高,且实验周期长、人力资源耗费大.
而新一代测序需要对大量外显子进行测序,进而研究疾病相关区域并SNP验证,传统的方法不能满足这一要求,在此背景下,NimbleGen公司和安捷伦相继开发了新的技术以解决这一重大问题,为全基因组外显子测序的广泛运用提供了平台.
2.
1.
1NimbleGen公司的外显子捕获芯片罗氏NimbleGen推出的基于HD2平台2.
1M外显子序列捕获芯片[6](SequenceCapture2.
1MHumanExomemicroarrays)主要是利用杂交和DNA微阵列技术基因分离原理来捕获目标区域.
其大致流程是将打断后的基因组DNA与定制的序列捕获芯片杂交,洗去未杂交上的片段,随后将富集的目标片段洗脱扩增,最后进行高通量测序.
该方法具有高特异性和高覆盖度、捕获区域可按需设计和省时省力等优点.
自2008年罗氏推出NimbleGen序列捕获2.
1M外显子组芯片以来,全球的研究人员已通过此款序列捕获芯片对成千上万的外显子组进行测序.
罗氏随之在2009年底推出的EZ外显子组序列捕获能提供液相的工作流程并以DNA液相探针杂交来捕获目的片段,该技术能提供高质量的目标序列富集以及更为灵活的可扩容性.
2.
1.
2安捷伦SureSelect靶向序列捕获系统安捷伦的外显子捕获系统[7]是基于寡核苷酸合成技术的液相靶向序列捕获系统.
其大致流程是将打断后的基因组DNA与SureSelect诱饵共孵,通过含有链酶亲和素标记的磁珠,钓出RNA诱饵-DNA第8期张鑫等:全基因组外显子测序及其应用849杂合体,洗脱磁珠,降解RNA诱饵,富集目的区域,再进行高通量测序.
基于液相序列捕获的高通量平行靶向测序技术具有高度特异性、高准确性、卓越的重现性和广泛的应用前景.
2.
2测序技术2.
2.
1传统的测序技术最早的DNA测序技术是1977年Sanger的"双脱氧链末端终止法"和Maxam及Gilbert的"化学降解法".
运用最多的是Sanger测序法,但这种测序仪一般一次最多只能同时进行96个或384个样品的测序.
Sanger测序技术经过30年的不断发展与完善,现在的测序长度可达1000bp,每一个碱基的读取准确率高达99.
999%,费用大约为0.
5美元/1000个碱基,已成为测序的金标准[8].
2.
2.
2新一代测序技术(Next-generationsequencing)传统的Sanger测序法无法大幅度的降低测序费用,而基因变异、RNA表达、蛋白与DNA相互作用以及染色体结构等研究需要使用高通量DNA测序技术.
而其他学科技术如计算机技术、聚合酶工程技术、数据存储分析技术的发展,均促使了新一代测序技术的进步.
新一代测序技术原理是:片断化的基因组DNA两侧连上接头,用不同的方法产生几百万个空间固定的PCR单克隆阵列,所有单克隆同时、独立地进行引物杂交和酶延伸反应,同时拍摄每个延伸所掺入的荧光标记信号来获取测序数据.
新一代测序技术广泛运用于Denovo测序、重测序、细菌基因组和比较基因组研究、小RNA测序、古生物学和古DNA研究领域以及环境基因组学和感染性疾病等研究领域.
近期市面上出现很多新一代测序仪产品,例如美国Illumina公司和英国Solexatechnology公司合作开发的Illumina测序仪[9]、美国RocheAppliedScience公司的454基因组测序仪[10]、美国AppliedBiosystems公司的SOLiD测序仪[11]、Dover/Harvard公司的Polonator测序仪以及美国Helicos公司的HeliScope单分子测序仪[12].
这些新型测序仪属于合成测序,大多使用循环芯片测序法(cyclic-arraysequencing),通过对布满DNA样品的芯片重复进行基于DNA的聚合酶反应(模板变性、引物复性杂交及延伸)以及荧光序列读取反应.
但其中单分子测序仪不需要扩增建立DNA库,而是基于边合成边测序的思想,将待测序列随机打断成小片段并在3′末端加上PolyA,通过合成互补链技术对数百万个DNA片段进行测序.
2.
3数据统计分析不同的新一代测序仪都能产生大量的数据,但如何在如此庞大的数据中分析出有意义的内容对于新一代测序来说无疑是一个巨大的挑战.
数据统计分析主要包括基本的数据分析(如图像的去噪、锐化、定位和偏移校正、依据光强度获得碱基)和生物信息分析(如检验靶区域的测序深度和覆盖度、比对序列、检测和注释SNPs和短小的插入或缺失(Shortinsertion/deletions,indels)),这对于测序数据的深入发掘具有重要的意义.
3全基因组外显子测序的相关研究3.
1在正常人中的相关研究Ng等[13]对一个体的全基因组外显子进行测序,结果发现10389个非同义SNPs(nsSNPs),其中5604个是杂合子,4785个是纯合子,大部分变异属于常见变异,且为中性.
同时检查了与疾病相关的已知基因,未发现该个体存在患某种重大疾病的风险.
这是首次对单个个体的外显子进行研究,不仅丰富了NCBI数据库的参考序列,而且为大规模的外显子测序提供了较具体的研究方法.
Li等[14]利用NimbleGen2.
1M芯片捕获外显子,GenomeAnalyzerII测序系统对200个丹麦人测序,在群体中共发现了121870个SNPs,包括53081个编码区的SNP位点(cSNP).
通过统计学方法对群体数据进行SNP的鉴定和等位基因频率的估算,获得了MAF在2%以上的cSNP的频率图谱;发现MAF为2%~5%的低频率区域,稀有的有害非同义cSNP是同义cSNP的1.
8倍.
从而间接说明了全基因组外显子测序能发现低频突变.
3.
2在孟德尔疾病和罕见综合症中的运用利用连锁分析方法研究孟德尔疾病需要足够多的患者、多代遗传的大家系,因此不适合难以收集到大家系和只有散发病例的孟德尔疾病,而全基因组外显子测序没有这些限制.
为验证外显子测序在孟德尔850HEREDITAS(Beijing)2011第33卷疾病研究中的可行性,Ng等[15]利用安捷伦244K芯片捕获外显子,GenomeAnalyzerII系统对12例研究对象进行测序,其中8例为HapMap计划中的个体,4例为无亲缘关系的弗里曼谢尔登综合征(Freeman-sheldonsyndrome,FSS)患者,FSS是一种由MYH3基因突变引起的常染色体显性遗传病.
同HapMap数据、全基因组测序数据(NA18507)和Sanger测序结果的比较表明了外显子测序数据的可靠性和研究遗传变异的敏感性与特异性.
通过逐步滤过法,同HapMap数据库和dbSNP数据库比对滤掉普遍变异和个人独具的变异后,验证了FSS的致病基因为MYH3.
以上研究表明,全基因组外显子测序可用于研究少数散发病例和有小家系的孟德尔疾病,为后续的研究提供了基于全基因组外显子测序策略的研究思路.
目前全基因组外显子测序除已广泛运用于孟德尔疾病的研究中,也能运用于一些代谢性疾病的研究.
依据全基因组外显子测序研究对象情况以及是否有定位区域,将目前的研究现状进行了总结(表1).
我国在该方面的研究也取得一定的成绩:2010年中南大学湘雅医院研究发现小脑共济失调新的致病基因TGM6[16],这是我国科学家应用外显子组测序技术进行孟德尔疾病研究的一项突破,对促进国内孟德尔疾病研究的发展具有重要意义.
随后通过该技术发现并验证了NCSTN基因的突变可导致逆向性痤疮的发生,对NCSTN基因突变的检测和逆向性痤疮及其相关疾病的诊断、治疗具有十分重要的意义[17].
表1全基因组外显子测序研究孟德尔疾病或综合征文献全基因组外显子测序疾病遗传模式致病基因捕获芯片测序平台1.
散发病例弗里曼谢尔登综合征[15]ADMYH3SureSelectHumanAllExonkitIlluminaGAIIKabuki综合征[18]ADMLL2SureSelectHumanAllExonkitIlluminaGAIISchinzel-Giedion综合征[19]ARSETBP1SureSelectHumanAllExonkitSOLiDSensenbrenner综合征[20]ARWDR35SureSelectHumanAllExonkitSOLiDFowler综合征[21]ARFLVCR2SureSelectHumanAllExonkitIlluminaGAIIXPerrault综合征[22]ARHSD17B4SureSelectHumanAllExonkitIlluminaGAIIXHajdu-Cheney综合征[23]ADNOTCH2SureSelectHumanAllExonkitIlluminaGAIIX成骨不全[24]ARSERPINF1SureSelectHumanAllExonkitSOLiD复合物I缺乏症[25]代谢性疾病ACAD9未提及SOLiD2.
散发和家系内病例米勒综合征[26]ARDHODHSureSelectHumanAllExonkitIlluminaGAIIBrown-Vialetto-vanLaere综合征[27]ARC20orf54NimbleGen2.
1MarrayIlluminaGAIIX3.
家系内病例血磷酸脂酶过多智力迟钝综合征[28]ARPIGVSureSelectHumanAllExonkitSOLiD家族性β-脂蛋白过少血症[29]ADANGPTL3SureSelectHumanAllExonkitIlluminaGAIIX色素性视网膜炎[30]ARDHDDSNimbleGen2.
1MarrayIlluminaGAIIX4.
家系内病例结合定位区域非综合征性耳聋[31]ARGPSM2SureSelectHumanAllExonkitIlluminaGAIIX肾脏相关性ciliopathy(NPHP-RC)[32]ARSDCCAG8NimbleGen385Karray*IlluminaGAIICarnevale,Malpuech,Michels和OSA综合征[33]ARMASP1SureSelectHumanAllExonkitIlluminaGAIIX原发性淋巴管性水肿[34]ADGJC2SureSelectHumanAllExonkitIlluminaGAIIX肌萎缩性侧索硬化(ALS)[35]ADVCPSureSelectHumanAllExonkitIlluminaGAIIX非综合征的智力迟钝[36]ARTECRAgilent244KmicroarrayIlluminaGAIIVanDenEnde-Gupta综合征[37]ARSCARF2SureSelectHumanAllExonkitIlluminaGAIIX自身免疫性淋巴组织增生症(ALPS)[38]ARFADDSureSelectHumanAllExonkitIlluminaGAIIX小脑共济失调[16]**ADTGM6NimbleGen2.
1MarrayIlluminaGAII逆向性痤疮[17]**ADNCSTNSureSelectHumanAllExonkitIlluminaHiseq2000注:AD是常染色体隐性遗传模式,AR是常染色体隐性遗传模式;GA是GenomeAnalyzer的缩写;*表示候选基因定制芯片;**表示该研究是中国的研究成果.
第8期张鑫等:全基因组外显子测序及其应用8513.
3在复杂疾病中的应用全基因组外显子测序能准确地找到孟德尔遗传疾病的致病基因,那么外显子测序同样也可用于研究常见疾病.
目前已有利用全基因组外显子测序的方法结合或不结合连锁分析结果研究复杂疾病的案例,例如:胰岛素抵抗动脉粥样硬化家庭研究主要是研究遗传和环境因素对西班牙裔和非裔美国人的葡萄糖稳态和肥胖症的影响,Bowden等[39]利用安捷伦SureSelect外显子捕获系统和GenomeAnalyzerIIx系统对浆乙二腈水平无显著性差别的2个家系中3个患者进行测序,发现ADIPOQ基因的低频突变(1.
1%)G45R,能解释17%的西班牙裔美国人的血浆乙二腈水平,63%的家族存在该突变;脑皮质发育异常是一组大脑功能障碍的疾病,影响到神经细胞增殖、迁移、组织形成等,Bilguvar等[40]运用NimbleGen2.
1M芯片捕获外显子和GenomeAnalyzerII测序系统对1例患者测序,发现WDR62基因与脑皮质发育异常疾病相关,从而能解释一系列的严重皮质畸形的症状,包括小头畸形、巨脑回畸形质等,该研究提示外显子测序对具有遗传异质性和诊断亚型困难的疾病具有一定的帮助.
早期主要运用传统的Sanger测序方法研究肿瘤患者的外显子以期发现新的体细胞突变,但耗时耗经费,随着新一代测序技术的发展,加快了对肿瘤的研究步伐.
3.
3.
1肿瘤基因组计划国际肿瘤基因组在Nature上发表的专题报道"Internationalnetworkofcancergenomeprojects"中详细描述了国际肿瘤基因组协作研究计划启动的历程、研究目标、研究策略、技术规范、数据分析与交汇及伦理学等核心问题[41].
国际肿瘤基因组协作组(ICGC)对人类常见的50种肿瘤(或亚型)进行全基因组变异分析,明确肿瘤发生发展过程中基因型与表型变异的关系,为全面解析肿瘤的生物学特性与机制提供基础数据.
我国肿瘤学和基因组学的科学家携手组建了"中国肿瘤基因组研究协作组(CCGC)",启动了中国肿瘤基因组研究项目并选择我国常见高发肿瘤胃癌作为参与ICGC的研究任务,拟采用外显子测序和典型病例的全基因组测序的方案.
在过去的3年中,运用外显子测序的研究方法,研究人员在卡波西肉瘤、小细胞肺癌、非小细胞肺癌、Hodgkin淋巴瘤、急性髓性白血病、小叶基底乳癌、慢性淋巴细胞性白血病、骨癌、葡萄膜黑色素瘤等肿瘤的研究中取得了显著的成就[42~48].
目前我国在白血病、黑色素瘤、多发性内分泌腺瘤综合征/家族性甲状腺髓样癌的研究中也取得了一定的成绩[43,49,50].
3.
3.
2运用新一代测序技术研究肿瘤的案例新一代测序技术可以用于研究肿瘤DNA,以发现核苷酸序列的改变和染色体结构改变,例如Harbour等[48]利用NimbleGen2.
1M芯片捕获外显子,GenomeAnalyzerII测序系统对31例转移性葡萄膜黑色素瘤患者测序,在84%的转移性肿瘤中发现了BAP1基因的失活性体细胞突变.
肿瘤DNA序列存在染色体易位、倒位、重复和缺失,随着双向测序的出现,可以通过组装,确定DNA片段的物理距离的原理研究肿瘤DNA的结构变异,Stephen等[47]研究发现许多肿瘤中2%~3%的患者存在大量的基因组重排现象.
同时可以通过对原发灶、转移灶和异种移植物DNA测序,研究肿瘤的进展情况,例如对基底样乳腺癌的原发肿瘤和转移灶或异种移植物测序发现,在转移灶或异种移植物的突变来源于原发灶,但是会选择性扩增[51].
而小叶乳腺癌的转移灶的多数突变(约60%)不存在原发灶中[44].
4在基因诊断中的运用由于全基因组外显子测序技术在孟德尔疾病和复杂疾病研究中的优势,目前已有成功运用该技术进行基因诊断和分子诊断的报道.
Choi等[52]通过NimbleGen2.
1M芯片和Illumina测序系统,对疑似Bartter综合征的5例高加索人进行测序,结果发现其中1例患者的SLC26A3基因(为先天性氯腹泻的位点)的一个错义突变D652N,说明该患者是先天性氯腹泻,而不是Bartter综合征,其临床也支持这个诊断.
后期对39例疑似Bartter综合征和95例对照进行Sanger测序验证,发现其中5例患者是SLC26A3纯合子突变,且临床表现也符合先天性氯腹泻诊断.
上述研究表明全基因组外显子测序可以用于发现疾病相关的基因信息,利用测序信息结合临床特征可对相关遗传病做出正确的诊断,从而进一步证852HEREDITAS(Beijing)2011第33卷明全基因组外显子测序能运用于疾病的分子诊断.
除此之外,还有利用全基因组外显子测序的策略对其他临床上难诊断的疾病进行诊断的相关报道,如新生儿糖尿病、难治性炎性肠病和Charcot-Marie-Toothatrophy综合征的分子水平的诊断[53-55].
5展望目前,全基因组外显子测序已在孟德尔疾病或罕见综合征的研究中取得了重大突破,证实了全基因组外显子测序对鉴定孟德尔疾病或罕见综合征的致病基因是行之有效的;可以运用在复杂疾病的研究中,目前在肿瘤方面的研究较多,但由于该技术存在一些尚待完善的问题,导致全基因组外显子测序在常见疾病的研究进展缓慢,但随着技术的发展和后续分析手段的丰富,预期在不久的将来,外显子测序会广泛地运用到其他复杂疾病的研究中;也可以用于疾病的分子诊断,为临床一些难以诊断的疾病提供了一种新诊断思路.
因此,Biesecker等[56]在NatureGenetics发表评论说:外显子测序使得医学基因组学成为现实.
全基因组外显子测序也存在不足之处:(1)该技术对结构变异与非编码区变异的研究具有局限性,而结构变异和非编码区变异也可能与疾病相关,可通过全基因组测序研究非编码区变异,同时进行基因组组装,同基因序列对比后发现一些结构变异,如CNV、indels,此外还可以结合芯片研究检测CNV;(2)在目标区域的捕获时,存在捕获不均、捕获偏差等现象,可以通过增加测序深度,获得更多的序列信息进行统计分析,以尽可能的弥补这些偏差;(3)研究常见疾病的罕见突变时,需要庞大的样本量,也导致了测序的费用的升高;目前用于验证的基因分型的平台如Sequenom、TaqMan主要适用于研究常见变异,需要定制芯片或通过Sanger测序,但这些方法耗时和价格较昂贵;数据分析的方法仍然不够完善,尚待解决,难以从海量的数据中迅速发现具有重大价值的信息.
尽管如此,由于外显子是与疾病及表型相关的最具特征性的区域,并且迄今为止较难评价非编码区域对疾病的影响,所以在全基因组测序费用居高不下的今天,全基因组外显子测序仍然不失为一个很好的选择.
外显子测序为阐明疾病的发病机理提供了新的线索,在疾病的基因诊断和致病基因的研究方面有广阔的前景,为之后的功能研究奠定了理论基础,从而为临床应用铺平了道路.
参考文献(References):[1]KuCS,LoyEY,PawitanY,ChiaKS.
Thepursuitofge-nome-wideassociationstudies:wherearewenowJHumGenet,2010,55(4):195–206.
[2]McClellanJM,SusserE,KingMC.
Schizophrenia:acommondiseasecausedbymultiplerarealleles.
BrJPsychiatry,2007,190:194–199.
[3]McClellanJ,KingMC.
Geneticheterogeneityinhumandisease.
Cell,2010,141(2):210–217.
[4]BotsteinD,RischN.
Discoveringgenotypesunderlyinghumanphenotypes:pastsuccessesformendeliandisease,futureapproachesforcomplexdisease.
NatGenet,2003,33(3):228–237.
[5]StensonPD,BallEV,MortM,PhillipsAD,ShielJA,ThomasNST,AbeysingheS,KrawczakM,CooperDN.
Humangenemutationdatabase(HGMD):2003update.
HumMutat,2003,21(6):577–581.
[6]KahvejianA,QuackenbushJ,ThompsonJF.
WhatwouldyoudoifyoucouldsequenceeverythingNatBiotechnol,2008,26(10):1125–1133.
[7]GnirkeA,MelnikovA,MaguireJ,RogovP,LeProustEM,BrockmanW,FennellT,GiannoukosG,FisherS,RussC,GabrielS,JaffeDB,LanderES,NusbaumC.
Solutionhybridselectionwithultra–longoligonucleotidesformas-sivelyparalleltargetedsequencing.
NatBiotechnol,2009,27(2):182–189.
[8]ShendureJ,JiH.
Next–generationDNAsequencing.
NatBiotechnol,2008,26(10):1135–1145.
[9]HillierLW,MarthGT,QuinlanAR,DoolingD,FewellG,BarnettD,FoxP,GlasscockJI,HickenbothamM,HuangW,MagriniVJ,RichtRJ,SanderSN,StewartDA,Strom-bergM,TsungEF,WylieT,SchedlT,WilsonRK,MardisER.
Whole–genomesequencingandvariantdiscoveryinC.
elegans.
NatMethods,2008,5(2):183–188.
[10]DroegeM,HillB.
TheGenomeSequencerFLXSys-tem–longerreads,moreapplications,straightforwardbioinformaticsandmorecompletedatasets.
JBiotechnol,2008,136(1–2):3–10.
[11]HashimotoS,QuW,AhsanB,OgoshiK,SasakiA,Naka-taniY,LeeY,OgawaM,AmetaniA,SuzukiY,SuganoS,LeeCC,NutterRC,MorishitaS,MatsushimaK.
High–resolutionanalysisofthe5'–endtranscriptomeusinganextgenerationDNAsequencer.
PLoSOne,2009,4(1):e4108.
第8期张鑫等:全基因组外显子测序及其应用853[12]RothbergJM,LeamonJH.
Thedevelopmentandimpactof454sequencing.
NatBiotechnol,2008,26(10):1117–1124.
[13]NgPC,LevyS,HuangJQ,StockwellTB,WalenzBP,LiK,AxelrodN,BusamDA,StrausbergRL,VenterJC.
Ge-neticvariationinanindividualhumanexome.
PLoSGenet,2008,4(8):e1000160.
[14]LiYR,VinckenboschN,TianG,Huerta–SanchezE,JiangT,JiangH,AlbrechtsenA,AndersenG,CaoHZ,KorneliussenT,GrarupN,GuoYR,HellmanI,JinX,LiQB,LiuJT,LiuX,SparsoT,TangMF,WuHL,WuRH,YuC,ZhengHC,As-trupA,BolundL,HolmkvistJ,JrgensenT,KristiansenK,SchmitzO,SchwartzTW,ZhangXQ,LiRQ,YangHM,WangJ,HansenT,PedersenO,NielsenR,WangJ.
Rese-quencingof200humanexomesidentifiesanexcessoflow–frequencynon–synonymouscodingvariants.
NatGenet,2010,42(11):969–972.
[15]NgSB,TurnerEH,RobertsonPD,FlygareSD,BighamAW,LeeC,ShafferT,WongM,BhattacharjeeA,EichlerEE,BamshadM,NickersonDA,ShendureJ.
Targetedcaptureandmassivelyparallelsequencingof12humanexomes.
Na-ture,2009,461(7261):272–276.
[16]WangJL,YangX,XiaK,HuZM,WengL,JinX,JiangH,ZhangP,ShenL,GuoJF,LiN,LiYR,LeiLF,ZhouJ,DuJ,ZhouYF,PanQ,WangJ,WangJ,LiRQ,TangBS.
TGM6identifiedasanovelcausativegeneofspinocerebellarataxiasusingexomesequencing.
Brain,2010,133(Pt12):3510–3518.
[17]LiuY,GaoM,LvYM,YangX,RenYQ,JiangT,ZhangX,GuoBR,LiM,ZhangQ,ZhangP,ZhouFS,ChenG,YinXY,ZuoXB,SunLD,ZhengXD,ZhangSM,LiuJJ,ZhouYW,LiYR,WangJ,YangHM,YangS,LiRQ,ZhangXJ.
Con-firmationbyExomeSequencingofthePathogenicRoleofNCSTNMutationsinAcneInversa(HidradenitisSuppura-tiva).
JInvestDermatol,2011,131(7):1570–1572.
[18]NgSB,BighamAW,BuckinghamKJ,HannibalMC,McMil-linMJ,GildersleeveHI,BeckAE,TaborHK,CooperGM,MeffordHC,LeeC,TurnerEH,SmithJD,RiederMJ,Yo-shiuraKI,MatsumotoN,OhtaT,NiikawaN,NickersonDA,BamshadMJ,ShendureJ.
ExomesequencingidentifiesMLL2mutationsasacauseofKabukisyndrome.
NatGenet,2010,42(9):790–793.
[19]HoischenA,vanBonBW,GilissenC,ArtsP,vanLierB,SteehouwerM,deVriesP,deReuverR,WieskampN,Mor-tierG,DevriendtK,AmorimMZ,RevencuN,KiddA,Bar-bosaM,TurnerA,SmithJ,OleyC,HendersonA,HayesIM,ThompsonEM,BrunnerHG,deVriesBBA,VeltmanJA.
DenovomutationsofSETBP1causeSchinzel–Giedionsyn-drome.
NatGenet,2010,42(6):483–485.
[20]GilissenC,ArtsHH,HoischenA,SpruijtL,MansDA,ArtsP,vanLierB,SteehouwerM,vanReeuwijkJ,KantSG,Roep-manR,KnoersNVAM,VeltmanJA,BrunnerHG.
ExomesequencingidentifiesWDR35variantsinvolvedinSensen-brennersyndrome.
AmJHumGenet,2010,87(3):418–423.
[21]LalondeE,AlbrechtS,HaKCH,JacobK,BolducN,Poly-chronakosC,DechelotteP,MajewskiJ,JabadoN.
Unex-pectedallelicheterogeneityandspectrumofmutationsinFowlersyndromerevealedbynext–generationexomese-quencing.
HumMutat,2010,31(8):918–923.
[22]PierceSB,WalshT,ChisholmKM,LeeMK,ThorntonAM,FiumaraA,OpitzJM,Levy–LahadE,KlevitRE,KingMC.
MutationsintheDBP–deficiencyproteinHSD17B4causeovariandysgenesis,hearingloss,andataxiaofPerraultSyn-drome.
AmJHumGenet,2010,87(2):282–288.
[23]SimpsonMA,IrvingMD,AsilmazE,GrayMJ,DafouD,ElmslieFV,MansourS,HolderSE,BrainCE,BurtonBK,KimKH,PauliRM,AftimosS,StewartH,KimCA,Holder–EspinasseM,RobertsonSP,DrakeWM,TrembathRC.
MutationsinNOTCH2causeHajdu–Cheneysyndrome,adisorderofsevereandprogressiveboneloss.
NatGenet,2011,43(4):303–305.
[24]BeckerJ,SemlerO,GilissenC,LiY,BolzHJ,GiuntaC,BergmannC,RohrbachM,KoerberF,ZimmermannK,deVriesP,WirthB,SchoenauE,WollnikB,VeltmanJA,HoischenA,NetzerC.
Exomesequencingidentifiestruncat-ingmutationsinHumanSERPINF1inAutosomal–Recessiveosteogenesisimperfecta.
AmJHumGenet,2011,88(3):362–371.
[25]HaackTB,DanhauserK,HaberbergerB,HoserJ,StreckerV,BoehmD,UzielG,LamanteaE,InvernizziF,PoultonJ,RolinskiB,IusoA,BiskupS,SchmidtT,MewesHW,WittigI,MeitingerT,ZevianiM,ProkischH.
ExomesequencingidentifiesACAD9mutationsasacauseofcomplexIdefi-ciency.
NatGenet,2010,42(12):1131–1134.
[26]NgSB,BuckinghamKJ,LeeC,BighamAW,TaborHK,DentKM,HuffCD,ShannonPT,JabsEW,NickersonDA,Shen-dureJ,BamshadMJ.
Exomesequencingidentifiesthecauseofamendeliandisorder.
NatGenet,2009,42(1):30–35.
[27]JohnsonJO,GibbsJR,vanMaldergemL,HouldenH,Sin-gletonAB.
Exomesequencinginbrown–vialetto–vanlaeresyndrome.
AmJHumGenet,2010,87(4):567–569.
[28]KrawitzPM,SchweigerMR,RdelspergerC,MarcelisC,KlschU,MeiselC,StephaniF,KinoshitaT,MurakamiY,BauerS,IsauM,FischerA,DahlA,KerickM,HechtJ,KhlerS,JgerM,GrünhagenJ,deCondorBJ,DoelkenS,854HEREDITAS(Beijing)2011第33卷BrunnerHG,MeineckeP,PassargeE,ThompsonMD,ColeDE,HornD,RoscioliT,MundlosS,RobinsonPN.
Iden-tity–by–descentfilteringofexomesequencedataidentifiesPIGVmutationsinhyperphosphatasiamentalretardationsyndrome.
NatGenet,2010,42(10):827–829.
[29]MusunuruK,PirruccelloJP,DoR,PelosoGM,GuiducciC,SougnezC,GarimellaKV,FisherS,AbreuJ,BarryAJ,FennellT,BanksE,AmbrogioL,CibulskisK,KernytskyA,GonzalezE,RudziczN,EngertJC,DepristoMA,DalyMJ,CohenJC,HobbsHH,AltshulerD,SchonfeldG,GabrielSB,YueP,KathiresanS.
Exomesequencing,ANGPTL3muta-tions,andfamilialcombinedHypolipidemia.
NEnglJMed,2010,363(23):2220–2227.
[30]ZüchnerS,DallmanJ,WenR,BeechamG,NajA,FarooqA,KohliMA,WhiteheadPL,HulmeW,KonidariI,EdwardsYJK,CaiGQ,PeterI,SeoD,BuxbaumJD,HainesJL,BlantonS,YoungJ,AlfonsoE,VanceJM,LamBL,Peri-cak–VanceMA.
Whole–ExomesequencinglinksavariantinDHDDStoretinitispigmentosa.
AmJHumGenet,2011,88(2):201–206.
[31]WalshT,ShahinH,Elkan–MillerT,LeeMK,ThorntonAM,RoebW,AbuRayyanA,LoulusS,AvrahamKB,KingMC,KanaanM.
WholeexomesequencingandhomozygositymappingidentifymutationinthecellpolarityproteinGPSM2asthecauseofnonsyndromichearinglossDFNB82.
AmJHumGenet,2010,87(1):90–94.
[32]OttoEA,HurdTW,AirikR,ChakiM,ZhouWB,StoetzelC,PatilSB,LevyS,GhoshAK,Murga–ZamalloaCA,vanReeuwijkJ,LetteboerSJF,SangLY,GilesRH,LiuQ,CoeneKLM,Estrada–CuzcanoA,CollinRWJ,McLaughlinHM,HeldS,KasanukiJM,RamaswamiG,ConteJ,LopezI,WashburnJ,MacDonaldJ,HuJH,YamashitaY,MaherER,Guay–WoodfordLM,NeumannHPH,ObermüllerN,KoenekoopRK,BergmannC,BeiXS,LewisRA,KatsanisN,LopesV,WilliamsDS,LyonsRH,DangCV,BritoDA,DiasMB,ZhangXM,CavalcoliJD,NürnbergG,NürnbergP,PierceEA,JacksonPK,AntignacC,SaunierS,RoepmanR,DollfusH,KhannaH,HildebrandtF.
Candidateexomecap-tureidentifiesmutationofSDCCAG8asthecauseofareti-nal–renalciliopathy.
NatGenet,2010,42(10):840–850.
[33]SirmaciA,WalshT,AkayH,SpiliopoulosM,akalarYB,Hasanefendiolu–BayrakA,DumanD,FarooqA,KingMC,TekinM.
MASP1mutationsinpatientswithfacial,umbilical,coccygeal,andauditoryfindingsofcarnevale,malpuech,OSA,andmichelssyndromes.
AmJHumGenet,2010,87(5):679–686.
[34]OstergaardP,SimpsonMA,BriceG,MansourS,ConnellFC,OnoufriadisA,ChildAH,HwangJ,KalidasK,MortimerPS,TrembathR,JefferyS.
RapididentificationofmutationsinGJC2inprimarylymphoedemausingwholeexomesequenc-ingcombinedwithlinkageanalysiswithdelineationofthephenotype.
JMedGenet,2011,48(4):251–255.
[35]JohnsonJO,MandrioliJ,BenatarM,AbramzonY,vanDeer-linVM,TrojanowskiJQ,GibbsJR,BrunettiM,GronkaS,WuuJ,DingJH,McCluskeyL,Martinez–LageM,FalconeD,HernandezDG,ArepalliS,ChongS,SchymickJC,RothsteinJ,LandiF,WangYD,CalvoA,MoraG,SabatelliM,Mon-surròMR,BattistiniS,SalviF,SpataroR,SolaP,BorgheroG,GalassiG,ScholzSW,TaylorJP,RestagnoG,ChioA,TraynorBJ.
ExomesequencingrevealsVCPmutationsasacauseoffamilialALS.
Neuron,2010,68(5):857–864.
[36]CaliskanM,ChongJX,UricchioL,AndersonR,ChenP,SougnezC,GarimellaK,GabrielSB,dePristoMA,ShakirK,MaternD,DasS,WaggonerD,NicolaeDL,OberC.
Exomesequencingrevealsanovelmutationforautosomalrecessivenon–syndromicmentalretardationintheTECRgeneonchromosome19p13.
HumMolGenet,2010,20(7):1285–1289.
[37]AnastasioN,Ben–OmranT,TeebiA,HaKCH,LalondeE,AliR,AlmureikhiM,DerKaloustianVM,LiuJH,RosenblattDS,MajewskiJ,Jerome–MajewskaLA.
MutationsinSCARF2areresponsibleforVanDenEnde–Guptasyndrome.
AmJHumGenet,2010,87(4):553–559.
[38]BolzeA,ByunM,McDonaldD,MorganNV,AbhyankarA,PremkumarL,PuelA,BaconCM,Rieux–LaucatF,PangK,BritlandA,AbelL,CantA,MaherER,RiedlSJ,HambletonS,CasanovaJL.
Whole-Exome-Sequencing-BaseddiscoveryofHumanFADDdeficiency.
AmJHumGenet,2010,87(6):873–881.
[39]BowdenDW,AnSS,PalmerND,BrownWM,NorrisJM,HaffnerSM,HawkinsGA,GuoX,RotterJI,ChenYD,WagenknechtLE,LangefeldCD.
Molecularbasisofalink-agepeak:exomesequencingandfamily–basedanalysisiden-tifyararegeneticvariantintheADIPOQgeneintheIRASFamilyStudy.
HumMolGenet,201019(20):4112–4120.
[40]BilgüvarK,zturkAK,LouviA,KwanKY,ChoiM,TatliB,YalnizoluD,TüysüzB,alayanAO,GkbenS,Kay-makalanH,BarakT,BakircioluM,YasunoK,HoW,SandersS,ZhuY,YilmazS,DinerA,JohnsonMH,BronenRA,KoerN,PerH,ManeS,PamirMN,YalinkayaC,Ku-mandaS,TopuM,zmenM,estanN,LiftonRP,StateMW,GünelM.
Whole–exomesequencingidentifiesrecessiveWDR62mutationsinseverebrainmalformations.
Nature,2010,467(7312):207–210.
第8期张鑫等:全基因组外显子测序及其应用855[41]HudsonTJ,AndersonW,ArtezA,BarkerAD,BellC,Bern-abéRR,BhanMK,CalvoF,EerolaI,GerhardDS,Gutt-macherA,GuyerM,HemsleyFM,JenningsJL,KerrD,KlattP,KolarP,KusadaJ,LaneDP,LaplaceF,YouyongL,Net-tekovenG,OzenbergerB,PetersonJ,RaoTS,RemacleJ,SchaferAJ,ShibataT,StrattonMR,VockleyJG,WatanabeK,YangH,YuenMM,KnoppersBM,BobrowM,Cam-bon–ThomsenA,DresslerLG,DykeSO,JolyY,KatoK,KennedyKL,NicolásP,ParkerMJ,Rial–SebbagE,Ro-meo–CasabonaCM,ShawKM,WallaceS,WiesnerGL,ZepsN,LichterP,BiankinAV,ChabannonC,ChinL,ClémentB,deAlavaE,DegosF,FergusonML,GearyP,HayesDN,HudsonTJ,JohnsAL,KasprzykA,NakagawaH,PennyR,PirisMA,SarinR,ScarpaA,vandeVijverM,FutrealPA,AburataniH,BayésM,BotwellDD,CampbellPJ,EstivillX,GerhardDS,GrimmondSM,GutI,HirstM,López–OtínC,MajumderP,MarraM,McPhersonJD,NakagawaH,NingZ,PuenteXS,RuanY,ShibataT,StrattonMR,StunnenbergHG,SwerdlowH,VelculescuVE,WilsonRK,XueHH,YangL,SpellmanPT,BaderGD,BoutrosPC,CampbellPJ,FlicekP,GetzG,GuigóR,GuoG,HausslerD,HeathS,HubbardTJ,JiangT,JonesSM,LiQ,López–BigasN,LuoR,Mut-huswamyL,OuelletteBF,PearsonJV,PuenteXS,QuesadaV,RaphaelBJ,SanderC,ShibataT,SpeedTP,SteinLD,StuartJM,TeagueJW,TotokiY,TsunodaT,ValenciaA,WheelerDA,WuH,ZhaoS,ZhouG,SteinLD,GuigóR,HubbardTJ,JolyY,JonesSM,KasprzykA,LathropM,López–BigasN,OuelletteBF,SpellmanPT,TeagueJW,ThomasG,ValenciaA,YoshidaT,KennedyKL,AxtonM,DykeSO,FutrealPA,GerhardDS,GunterC,GuyerM,HudsonTJ,McPhersonJD,MillerLJ,OzenbergerB,ShawKM,KasprzykA,SteinLD,ZhangJ,HaiderSA,WangJ,YungCK,CrossA,LiangY,GnaneshanS,GubermanJ,HsuJ,BobrowM,ChalmersDR,HaselKW,JolyY,KaanTS,KennedyKL,KnoppersBM,LowranceWW,MasuiT,NicolásP,Rial–SebbagE,Rodri-guezLL,VergelyC,YoshidaT,GrimmondSM,BiankinAV,BowtellDD,CloonanN,deFazioA,EshlemanJR,Etemad-moghadamD,GardinerBA,KenchJG,ScarpaA,SutherlandRL,TemperoMA,WaddellNJ,WilsonPJ,McPhersonJD,GallingerS,TsaoMS,ShawPA,PetersenGM,Muk-hopadhyayD,DePinhoRA,ThayerS,ShazandK,BeckT,SamM,TimmsL,BallinV,LuY,JiJ,ZhangX,ChenF,HuX,ZhouG,YangQ,TianG,ZhangL,XingX,LiX,ZhuZ,YuY,YuJ,YangH,LathropM,TostJ,BrennanP,HolcatovaI,ZaridzeD,BrazmaA,EgevardL,ProkhortchoukE,BanksRE,UhlénM,Cambon–ThomsenA,ViksnaJ,PontenF,SkryabinK,StrattonMR,FutrealPA,BirneyE,BorgA,Borresen–DaleAL,CaldasC,FoekensJA,MartinS,Reis–FilhoJS,RichardsonAL,SotiriouC,StunnenbergHG,ThomsG,vandeVijverM,van'tVeerL,CalvoF,BirnbaumD,BlancheH,BoucherP,BoyaultS,ChabannonC,GutI,Masson–JacquemierJD,LathropM,PauportéI,PivotX,Vincent–SalomonA,TaboneE,TheilletC,ThomasG,TostJ,TreilleuxI,Bioulac–SageP,ClémentB,DecaensT,DegosF,FrancoD,GutI,GutM,HeathS,LathropM,SamuelD,ThomasG,Zucman–RossiJ,LichterP,EilsR,BrorsB,KorbelJO,KorshunovA,LandgrafP,LehrachH,PfisterS,RadlwimmerB,ReifenbergerG,TaylorMD,vonKalleC,MajumderPP,SarinR,RaoTS,BhanMK,ScarpaA,Peder-zoliP,LawlorRA,DelledonneM,BardelliA,BiankinAV,GrimmondSM,GressT,KlimstraD,ZamboniG,ShibataT,NakamuraY,NakagawaH,KusadaJ,TsunodaT,MiyanoS,AburataniH,KatoK,FujimotoA,YoshidaT,CampoE,López–OtínC,EstivillX,GuigóR,deSanjoséS,PirisMA,MontserratE,González–DíazM,PuenteXS,JaresP,Valen-ciaA,HimmelbaueH,QuesadaV,BeaS,StrattonMR,Fut-realPA,CampbellPJ,Vincent–SalomonA,RichardsonAL,Reis–FilhoJS,vandeVijverM,ThomasG,Mas-son–JacquemierJD,AparicioS,BorgA,Brresen–DaleAL,CaldasC,FoekensJA,StunnenbergHG,van'tVeerL,EastonDF,SpellmanPT,MartinS,BarkerAD,ChinL,CollinsFS,ComptonCC,FergusonML,GerhardDS,GetzG,GunterC,GuttmacherA,GuyerM,HayesDN,LanderES,OzenbergerB,PennyR,PetersonJ,SanderC,ShawKM,SpeedTP,SpellmanPT,VockleyJG,WheelerDA,WilsonRK,HudsonTJ,ChinL,KnoppersBM,LanderES,LichterP,SteinLD,StrattonMR,AndersonW,BarkerAD,BellC,BobrowM,BurkeW,CollinsFS,ComptonCC,DePinhoRA,EastonDF,FutrealPA,GerhardDS,GreenAR,GuyerM,HamiltonSR,HubbardTJ,KallioniemiOP,KennedyKL,LeyTJ,LiuET,LuY,MajumderP,MarraM,OzenbergerB,PetersonJ,SchaferAJ,SpellmanPT,StunnenbergHG,WainwrightBJWilsonRK,YangH.
Internationalnetworkofcancergenomeprojects.
Nature,2010,464(7291):993–998.
[42]ByunM,AbhyankarA,LelargeV,PlancoulaineS,PalanduzA,TelhanL,BoissonB,PicardC,DewellS,ZhaoCN,JouanguyE,FeskeS,AbelL,CasanovaJL.
Whole–exomesequencing–baseddiscoveryofSTIM1deficiencyinachildwithfatalclassicKaposisarcoma.
JExpMed,2010,207(11):2307–2312.
[43]YanXJ,XuJ,GuZH,PanCM,LuG,ShenY,ShiJY,ZhuYM,TangL,ZhangXW,LiangWX,MiJQ,SongHD,LiKQ,ChenZ,ChenSJ.
ExomesequencingidentifiessomaticmutationsofDNAmethyltransferasegeneDNMT3Ainacute856HEREDITAS(Beijing)2011第33卷monocyticleukemia.
NatGenet,2011,43(4):309–315.
[44]ShahSP,MorinRD,KhattraJ,PrenticeL,PughT,BurleighA,DelaneyA,GelmonK,GulianyR,SenzJ,SteidlC,HoltRA,JonesS,SunM,LeungG,MooreR,SeversonT,TaylorGA,TeschendorffAE,TseK,TurashviliG,VarholR,WarrenRL,WatsonP,ZhaoYJ,CaldasC,HuntsmanD,HirstM,MarraMA,AparicioS.
Mutationalevolutioninalobularbreasttumourprofiledatsinglenucleotideresolution.
Nature,2009,461(7265):809–813.
[45]LeeW,JiangZS,LiuJF,HavertyPM,GuanYH,StinsonJ,YueP,ZhangY,PantKP,BhattD,HaC,JohnsonS,Kenne-merMI,MohanS,NazarenkoI,WatanabeC,SparksAB,ShamesDS,GentlemanR,deSauvageFJ,SternH,PanditaA,BallingerDG,DrmanacR,ModrusanZ,SeshagiriS,ZhangZM.
Themutationspectrumrevealedbypairedgenomese-quencesfromalungcancerpatient.
Nature,2010,465(7297):473–477.
[46]SaarinenS,AavikkoM,AittomkiK,LaunonenV,LehtonenR,FranssilaK,LehtonenHJ,KaasinenE,BroderickP,Tark-kanenJ,BainBJ,BauduerF,UnalA,SwerdlowAJ,CookeR,MakinenMJ,HoulstonR,VahteristoP,AaltonenLA.
ExomesequencingrevealsgermlineNPATmutationasacandidateriskfactorforHodgkinlymphoma.
Blood,2011.
[47]StephensPJ,GreenmanCD,FuBY,YangFT,BignellGR,MudieLJ,PleasanceED,LauKW,BeareD,StebbingsLA,McLarenS,LinML,McBrideDJ,VarelaI,Nik–ZainalS,LeroyC,JiaMM,MenziesA,ButlerAP,TeagueJW,QuailMA,BurtonJ,SwerdlowH,CarterNP,MorsbergerLA,Iacobuzio–DonahueC,FollowsGA,GreenAR,FlanaganAM,StrattonMR,FutrealPA,CampbellPJ.
Massivege-nomicrearrangementacquiredinasinglecatastrophiceventduringcancerdevelopment.
Cell,2011,144(1):27–40.
[48]HarbourJW,OnkenMD,RobersonEDO,DuanSH,CaoL,WorleyLA,CouncilML,MatatallKA,HelmsC,BowcockAM.
FrequentmutationofBAP1inmetastasizinguvealmelanomas.
Science,2010,330(6009):1410–1413.
[49]WeiXM,WaliaV,LinJC,TeerJK,PrickettTD,GartnerJ,DavisS,Stemke–HaleK,DaviesMA,GershenwaldJE,RobinsonW,RobinsonS,RosenbergSA,SamuelsY.
ExomesequencingidentifiesGRIN2Aasfrequentlymutatedinmelanoma.
NatGenet,2011,43(5):442–446.
[50]QiXP,MaJM,DuZF,YingRB,FeiJ,JinHY,HanJS,WangJQ,ChenXL,ChenCY,LiuWT,LuJJ,ZhangJG,ZhangXN.
RETgermlinemutationsidentifiedbyexomesequencinginaChinesemultipleendocrineneoplasiaType2A/Familialme-dullarythyroidcarcinomafamily.
PLoSOne,2011,6(5):e20353.
[51]DingL,EllisMJ,LiSQ,LarsonDE,ChenK,WallisJW,HarrisCC,McLellanMD,FultonRS,FultonLL,AbbottRM,HoogJ,DoolingDJ,KoboldtDC,SchmidtH,KalickiJ,ZhangQY,ChenL,LinL,WendlMC,McMichaelJF,Ma-griniVJ,CookL,McGrathSD,VickeryTL,AppelbaumE,DeschryverK,DaviesS,GuintoliT,LinL,CrowderR,TaoY,SniderJE,SmithSM,DukesAF,SandersonGE,PohlCS,DelehauntyKD,FronickCC,PapeKA,ReedJS,RobinsonJS,HodgesJS,SchierdingW,DeesND,ShenD,LockeDP,WiechertME,EldredJM,PeckJB,OberkfellBJ,LolofieJT,DuFY,HawkinsAE,O'LaughlinMD,BernardKE,Cun-ninghamM,ElliottG,MasonMD,ThompsonDMJr,Ivano-vichJL,GoodfellowPJ,PerouCM,WeinstockGM,AftR,WatsonM,LeyTJ,WilsonRK,MardisER.
Genomeremod-ellinginabasal–likebreastcancermetastasisandxenograft.
Nature,2010,464(7291):999–1005.
[52]ChoiM,SchollUI,JiWZ,LiuTW,TikhonovaIR,ZumboP,NayirA,BakkaloluA,zenS,SanjadS,Nelson–WilliamsC,FarhiA,ManeS,LiftonRP.
GeneticdiagnosisbywholeexomecaptureandmassivelyparallelDNAsequencing.
ProcNatlAcadSciUSA,2009,106(45):19096–19101.
[53]WortheyEA,MayerAN,SyversonGD,HelblingD,BonacciBB,DeckerB,SerpeJM,DasuT,TschannenMR,VeithRL,BasehoreMJ,BroeckelU,Tomita–MitchellA,ArcaMJ,CasperJT,MargolisDA,BickDP,HessnerMJ,RoutesJM,VerbskyJW,JacobHJ,DimmockDP.
Makingadefinitivediagnosis:Successfulclinicalapplicationofwholeexomesequencinginachildwithintractableinflammatoryboweldisease.
GenetMed,2010,13(3):255–262.
[54]BonnefondA,DurandE,SandO,DeGraeveF,GallinaS,BusiahK,LobbensS,SimonA,Bellanné–ChantelotC,LétourneauL,ScharfmannR,DelplanqueJ,SladekR,PolakM,VaxillaireM,FroguelP.
Moleculardiagnosisofneonataldiabetesmellitususingnext–generationsequencingofthewholeexome.
PLoSOne,2010,5(10):e13630.
[55]MontenegroG,PowellE,HuangJ,SpezianiF,EdwardsYJK,BeechamG,HulmeW,SiskindC,VanceJ,ShyM,ZüchnerS.
ExomesequencingallowsforrapidgeneidentificationinaCharcot–Marie–Toothfamily.
AnnNeurol,2011,69(3):464–470.
[56]BieseckerLG.
Exomesequencingmakesmedicalgenomicsareality.
NatGenet,42(1):13–14.
chinagene.
cn综述收稿日期:20000000;修回日期:20000000基金项目:国家高技术研究发展计划项目(863计划)(编号:2007AA02Z161)资助作者简介:张鑫,博士,研究方向:复杂疾病遗传学.
E-mail:zhangxin68619@163.
com通讯作者:张学军,博士,主任医师,教授,博士生导师,研究方向:复杂疾病遗传学.
E-mail:ayzxj@vip.
sina.
comDOI:10.
3724/SP.
J.
1005.
2011.
00847全基因组外显子测序及其应用张鑫,李敏,张学军安徽医科大学皮肤病研究所,合肥230032摘要:近年来,众多研究小组开展了大量的全基因组关联研究(Genome-wideassociationstudies,GWAS),发现并鉴定了许多与复杂疾病/性状相关联的遗传变异,为复杂疾病发病机制的研究提供了重要线索.
由于GWAS的结果存在假阳性、假阴性、检测到的单核苷酸多态性很少位于功能区以及对稀有变异和结构变异不敏感等问题,导致了其应用的局限性.
而新一代测序技术的进步,促进了全基因组测序和全基因组外显子测序的快速发展,为解决上述问题提供了契机.
全基因组外显子测序是利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法.
由于其具有对常见和罕见变异高灵敏度,能发现外显子区绝大部分疾病相关变异以及仅需要对约1%的基因组进行测序等优点,促使全基因组外显子测序成为鉴定孟德尔疾病的致病基因最有效的策略,也被运用于复杂疾病易感基因的研究和临床诊断中.
关键词:外显子测序;孟德尔疾病;复杂疾病;临床诊断ExomesequencinganditsapplicationZHANGXin,LIMin,ZHANGXue-JunInstituteofDermatologyofAnhuiMedicalUniversity,Hefei230032,ChinaAbstract:Inrecentyears,researchershaveidentifiedalargenumberofcomplexdiseases/traits-associatedgeneticvari-antsbyperforminggenome-wideassociationstudies(GWAS),whichmayprovideimportantcluesonunderstandingthemechanismsofrelateddiseases.
However,GWAShasitsownlimitationsintermsofbeingfalsepositive,falsenegativeresults,veryfewSNPslocatedinthefunctionalareasandinsensitivetodetectrareandstructuralvariations,whichresultsintheapplicationlimitationofthismethod.
Withthedevelopmentofthenext-generationsequencingtechnology,wholege-nomeandexomesequencingdevelopedrapidlyandprovideanopportunityforustodealwiththeproblemcausedbyGWAS.
Thishigh-throughputsequencingtechnologyisappliedforsequencingtheexome(1%ofgenome)todiscovermostofthediseases-relatedvariationsinexons.
Furthermore,itishighlyeffectivetodetectcommonandrarevariations.
Duetotheseadvantages,exomesequencinghasbecomeapowerfulandefficientstrategyforidentifyingthegenesresponsibleformendeliandisordersandcomplexdiseases,whichwillbeveryhelpfulforthediseasesclinicaldiagnosis.
Keywords:exomesequencing;mendeliandisorders;complexdiseases;clinicaldiagnosis随着人类基因组计划(Humangenomeproject,HGP)和国际人类基因组单体型图计划(Theinterna-tionalHapMapproject)的完成以及高通量生物芯片技术的成功研发,人们广泛利用高通量全基因组生848HEREDITAS(Beijing)2011第33卷物芯片的技术手段,采用关联分析的方法即全基因组关联研究(Genome-wideassociationstudies,GWAS)来筛选复杂疾病易感基因,取得了前所未有的成就.
但由于GWAS存在着一定的局限性,同时随着新一代测序技术的迅猛发展,促进了全基因组测序和全基因组外显子测序的发展.
本文就全基因组外显子测序的研究方法及其应用作一综述.
1外显子测序的产生背景GWAS是通过对大规模的群体(病例/对照)DNA样本进行包括单核苷酸多态性变异(Single-nucleotidepolymorphisms,SNPs)、拷贝数变异(Copynumbervariation,CNV)在内的全基因组高密度遗传标记并分型,从而寻找与复杂疾病相关的遗传因素的研究方法,掀起了人类基因组研究第3次浪潮.
自2005年以来,利用GWAS对多种常见疾病进行了研究,发现和重复验证了近2000个SNPs或位点,其中包括以前未检测到的而与疾病密切相关的基因及部分未知基因[1].
但此方法有其欠缺,如容易产生假阳性和假阴性,而且发现的与疾病关联的SNPs多位于基因间或内含子上,很少位于功能区(如外显子区和5'UTR区).
同时,芯片检测位点有一定的有限性,除Illumina的新一代全基因组基因分型芯片外,多数是发现常见变异(Minorallelefrequency,MAF>5%),而对稀有变异(MAF<5%)和其他结构变异不敏感.
目前,GWAS是基于常见疾病/常见变异的假说,而越来越多的研究结果表明许多复杂疾病是由稀有变异造成的[2,3],而这种基于芯片的GWAS在实验设计时尚未充分考虑这部分信息,故而较难搜寻稀有变异.
过去主要通过定位克隆、物理作图和候选基因测序的方法研究孟德尔疾病,但这些方法受到诸多因素的限制,如患者人数少、家系少或小、有意识的减少生育等,许多孟德尔疾病尚未发现其致病基因.
现有的技术和研究方法在研究孟德尔疾病和复杂疾病时均遇见了难以解决的问题,迫切需要新技术的出现.
而随着新一代测序技术的迅速发展、测序费用降低和时间缩短,使得全基因组和全基因组外显子测序在大规模人群中的运用成为现实.
由于全基因组测序的费用在短时间内很难降到与外显子测序相当的价格,在经费一定的情况下,全基因组外显子测序更适合大批量样本研究以获得高深度的测序数据.
更重要的是,目前一致认为大部分功能变异都潜藏在外显子中[4],这是因为引起孟德尔疾病的突变主要位于基因内造成的[4,5].
因此,全基因组外显子测序已成为现阶段基因测序工作的重心.
2外显子测序的基本思路及技术路线外显子测序主要包括目标区域序列的富集、DNA测序、生物信息学统计3个主要步骤.
2.
1目标区域序列的富集在过去的20年里,传统的Sanger测序主要是利用UniplexPCR和MutliplexPCR方法富集目标区域序列[6],但其引物合成与PCR反应所需费用都很高,且实验周期长、人力资源耗费大.
而新一代测序需要对大量外显子进行测序,进而研究疾病相关区域并SNP验证,传统的方法不能满足这一要求,在此背景下,NimbleGen公司和安捷伦相继开发了新的技术以解决这一重大问题,为全基因组外显子测序的广泛运用提供了平台.
2.
1.
1NimbleGen公司的外显子捕获芯片罗氏NimbleGen推出的基于HD2平台2.
1M外显子序列捕获芯片[6](SequenceCapture2.
1MHumanExomemicroarrays)主要是利用杂交和DNA微阵列技术基因分离原理来捕获目标区域.
其大致流程是将打断后的基因组DNA与定制的序列捕获芯片杂交,洗去未杂交上的片段,随后将富集的目标片段洗脱扩增,最后进行高通量测序.
该方法具有高特异性和高覆盖度、捕获区域可按需设计和省时省力等优点.
自2008年罗氏推出NimbleGen序列捕获2.
1M外显子组芯片以来,全球的研究人员已通过此款序列捕获芯片对成千上万的外显子组进行测序.
罗氏随之在2009年底推出的EZ外显子组序列捕获能提供液相的工作流程并以DNA液相探针杂交来捕获目的片段,该技术能提供高质量的目标序列富集以及更为灵活的可扩容性.
2.
1.
2安捷伦SureSelect靶向序列捕获系统安捷伦的外显子捕获系统[7]是基于寡核苷酸合成技术的液相靶向序列捕获系统.
其大致流程是将打断后的基因组DNA与SureSelect诱饵共孵,通过含有链酶亲和素标记的磁珠,钓出RNA诱饵-DNA第8期张鑫等:全基因组外显子测序及其应用849杂合体,洗脱磁珠,降解RNA诱饵,富集目的区域,再进行高通量测序.
基于液相序列捕获的高通量平行靶向测序技术具有高度特异性、高准确性、卓越的重现性和广泛的应用前景.
2.
2测序技术2.
2.
1传统的测序技术最早的DNA测序技术是1977年Sanger的"双脱氧链末端终止法"和Maxam及Gilbert的"化学降解法".
运用最多的是Sanger测序法,但这种测序仪一般一次最多只能同时进行96个或384个样品的测序.
Sanger测序技术经过30年的不断发展与完善,现在的测序长度可达1000bp,每一个碱基的读取准确率高达99.
999%,费用大约为0.
5美元/1000个碱基,已成为测序的金标准[8].
2.
2.
2新一代测序技术(Next-generationsequencing)传统的Sanger测序法无法大幅度的降低测序费用,而基因变异、RNA表达、蛋白与DNA相互作用以及染色体结构等研究需要使用高通量DNA测序技术.
而其他学科技术如计算机技术、聚合酶工程技术、数据存储分析技术的发展,均促使了新一代测序技术的进步.
新一代测序技术原理是:片断化的基因组DNA两侧连上接头,用不同的方法产生几百万个空间固定的PCR单克隆阵列,所有单克隆同时、独立地进行引物杂交和酶延伸反应,同时拍摄每个延伸所掺入的荧光标记信号来获取测序数据.
新一代测序技术广泛运用于Denovo测序、重测序、细菌基因组和比较基因组研究、小RNA测序、古生物学和古DNA研究领域以及环境基因组学和感染性疾病等研究领域.
近期市面上出现很多新一代测序仪产品,例如美国Illumina公司和英国Solexatechnology公司合作开发的Illumina测序仪[9]、美国RocheAppliedScience公司的454基因组测序仪[10]、美国AppliedBiosystems公司的SOLiD测序仪[11]、Dover/Harvard公司的Polonator测序仪以及美国Helicos公司的HeliScope单分子测序仪[12].
这些新型测序仪属于合成测序,大多使用循环芯片测序法(cyclic-arraysequencing),通过对布满DNA样品的芯片重复进行基于DNA的聚合酶反应(模板变性、引物复性杂交及延伸)以及荧光序列读取反应.
但其中单分子测序仪不需要扩增建立DNA库,而是基于边合成边测序的思想,将待测序列随机打断成小片段并在3′末端加上PolyA,通过合成互补链技术对数百万个DNA片段进行测序.
2.
3数据统计分析不同的新一代测序仪都能产生大量的数据,但如何在如此庞大的数据中分析出有意义的内容对于新一代测序来说无疑是一个巨大的挑战.
数据统计分析主要包括基本的数据分析(如图像的去噪、锐化、定位和偏移校正、依据光强度获得碱基)和生物信息分析(如检验靶区域的测序深度和覆盖度、比对序列、检测和注释SNPs和短小的插入或缺失(Shortinsertion/deletions,indels)),这对于测序数据的深入发掘具有重要的意义.
3全基因组外显子测序的相关研究3.
1在正常人中的相关研究Ng等[13]对一个体的全基因组外显子进行测序,结果发现10389个非同义SNPs(nsSNPs),其中5604个是杂合子,4785个是纯合子,大部分变异属于常见变异,且为中性.
同时检查了与疾病相关的已知基因,未发现该个体存在患某种重大疾病的风险.
这是首次对单个个体的外显子进行研究,不仅丰富了NCBI数据库的参考序列,而且为大规模的外显子测序提供了较具体的研究方法.
Li等[14]利用NimbleGen2.
1M芯片捕获外显子,GenomeAnalyzerII测序系统对200个丹麦人测序,在群体中共发现了121870个SNPs,包括53081个编码区的SNP位点(cSNP).
通过统计学方法对群体数据进行SNP的鉴定和等位基因频率的估算,获得了MAF在2%以上的cSNP的频率图谱;发现MAF为2%~5%的低频率区域,稀有的有害非同义cSNP是同义cSNP的1.
8倍.
从而间接说明了全基因组外显子测序能发现低频突变.
3.
2在孟德尔疾病和罕见综合症中的运用利用连锁分析方法研究孟德尔疾病需要足够多的患者、多代遗传的大家系,因此不适合难以收集到大家系和只有散发病例的孟德尔疾病,而全基因组外显子测序没有这些限制.
为验证外显子测序在孟德尔850HEREDITAS(Beijing)2011第33卷疾病研究中的可行性,Ng等[15]利用安捷伦244K芯片捕获外显子,GenomeAnalyzerII系统对12例研究对象进行测序,其中8例为HapMap计划中的个体,4例为无亲缘关系的弗里曼谢尔登综合征(Freeman-sheldonsyndrome,FSS)患者,FSS是一种由MYH3基因突变引起的常染色体显性遗传病.
同HapMap数据、全基因组测序数据(NA18507)和Sanger测序结果的比较表明了外显子测序数据的可靠性和研究遗传变异的敏感性与特异性.
通过逐步滤过法,同HapMap数据库和dbSNP数据库比对滤掉普遍变异和个人独具的变异后,验证了FSS的致病基因为MYH3.
以上研究表明,全基因组外显子测序可用于研究少数散发病例和有小家系的孟德尔疾病,为后续的研究提供了基于全基因组外显子测序策略的研究思路.
目前全基因组外显子测序除已广泛运用于孟德尔疾病的研究中,也能运用于一些代谢性疾病的研究.
依据全基因组外显子测序研究对象情况以及是否有定位区域,将目前的研究现状进行了总结(表1).
我国在该方面的研究也取得一定的成绩:2010年中南大学湘雅医院研究发现小脑共济失调新的致病基因TGM6[16],这是我国科学家应用外显子组测序技术进行孟德尔疾病研究的一项突破,对促进国内孟德尔疾病研究的发展具有重要意义.
随后通过该技术发现并验证了NCSTN基因的突变可导致逆向性痤疮的发生,对NCSTN基因突变的检测和逆向性痤疮及其相关疾病的诊断、治疗具有十分重要的意义[17].
表1全基因组外显子测序研究孟德尔疾病或综合征文献全基因组外显子测序疾病遗传模式致病基因捕获芯片测序平台1.
散发病例弗里曼谢尔登综合征[15]ADMYH3SureSelectHumanAllExonkitIlluminaGAIIKabuki综合征[18]ADMLL2SureSelectHumanAllExonkitIlluminaGAIISchinzel-Giedion综合征[19]ARSETBP1SureSelectHumanAllExonkitSOLiDSensenbrenner综合征[20]ARWDR35SureSelectHumanAllExonkitSOLiDFowler综合征[21]ARFLVCR2SureSelectHumanAllExonkitIlluminaGAIIXPerrault综合征[22]ARHSD17B4SureSelectHumanAllExonkitIlluminaGAIIXHajdu-Cheney综合征[23]ADNOTCH2SureSelectHumanAllExonkitIlluminaGAIIX成骨不全[24]ARSERPINF1SureSelectHumanAllExonkitSOLiD复合物I缺乏症[25]代谢性疾病ACAD9未提及SOLiD2.
散发和家系内病例米勒综合征[26]ARDHODHSureSelectHumanAllExonkitIlluminaGAIIBrown-Vialetto-vanLaere综合征[27]ARC20orf54NimbleGen2.
1MarrayIlluminaGAIIX3.
家系内病例血磷酸脂酶过多智力迟钝综合征[28]ARPIGVSureSelectHumanAllExonkitSOLiD家族性β-脂蛋白过少血症[29]ADANGPTL3SureSelectHumanAllExonkitIlluminaGAIIX色素性视网膜炎[30]ARDHDDSNimbleGen2.
1MarrayIlluminaGAIIX4.
家系内病例结合定位区域非综合征性耳聋[31]ARGPSM2SureSelectHumanAllExonkitIlluminaGAIIX肾脏相关性ciliopathy(NPHP-RC)[32]ARSDCCAG8NimbleGen385Karray*IlluminaGAIICarnevale,Malpuech,Michels和OSA综合征[33]ARMASP1SureSelectHumanAllExonkitIlluminaGAIIX原发性淋巴管性水肿[34]ADGJC2SureSelectHumanAllExonkitIlluminaGAIIX肌萎缩性侧索硬化(ALS)[35]ADVCPSureSelectHumanAllExonkitIlluminaGAIIX非综合征的智力迟钝[36]ARTECRAgilent244KmicroarrayIlluminaGAIIVanDenEnde-Gupta综合征[37]ARSCARF2SureSelectHumanAllExonkitIlluminaGAIIX自身免疫性淋巴组织增生症(ALPS)[38]ARFADDSureSelectHumanAllExonkitIlluminaGAIIX小脑共济失调[16]**ADTGM6NimbleGen2.
1MarrayIlluminaGAII逆向性痤疮[17]**ADNCSTNSureSelectHumanAllExonkitIlluminaHiseq2000注:AD是常染色体隐性遗传模式,AR是常染色体隐性遗传模式;GA是GenomeAnalyzer的缩写;*表示候选基因定制芯片;**表示该研究是中国的研究成果.
第8期张鑫等:全基因组外显子测序及其应用8513.
3在复杂疾病中的应用全基因组外显子测序能准确地找到孟德尔遗传疾病的致病基因,那么外显子测序同样也可用于研究常见疾病.
目前已有利用全基因组外显子测序的方法结合或不结合连锁分析结果研究复杂疾病的案例,例如:胰岛素抵抗动脉粥样硬化家庭研究主要是研究遗传和环境因素对西班牙裔和非裔美国人的葡萄糖稳态和肥胖症的影响,Bowden等[39]利用安捷伦SureSelect外显子捕获系统和GenomeAnalyzerIIx系统对浆乙二腈水平无显著性差别的2个家系中3个患者进行测序,发现ADIPOQ基因的低频突变(1.
1%)G45R,能解释17%的西班牙裔美国人的血浆乙二腈水平,63%的家族存在该突变;脑皮质发育异常是一组大脑功能障碍的疾病,影响到神经细胞增殖、迁移、组织形成等,Bilguvar等[40]运用NimbleGen2.
1M芯片捕获外显子和GenomeAnalyzerII测序系统对1例患者测序,发现WDR62基因与脑皮质发育异常疾病相关,从而能解释一系列的严重皮质畸形的症状,包括小头畸形、巨脑回畸形质等,该研究提示外显子测序对具有遗传异质性和诊断亚型困难的疾病具有一定的帮助.
早期主要运用传统的Sanger测序方法研究肿瘤患者的外显子以期发现新的体细胞突变,但耗时耗经费,随着新一代测序技术的发展,加快了对肿瘤的研究步伐.
3.
3.
1肿瘤基因组计划国际肿瘤基因组在Nature上发表的专题报道"Internationalnetworkofcancergenomeprojects"中详细描述了国际肿瘤基因组协作研究计划启动的历程、研究目标、研究策略、技术规范、数据分析与交汇及伦理学等核心问题[41].
国际肿瘤基因组协作组(ICGC)对人类常见的50种肿瘤(或亚型)进行全基因组变异分析,明确肿瘤发生发展过程中基因型与表型变异的关系,为全面解析肿瘤的生物学特性与机制提供基础数据.
我国肿瘤学和基因组学的科学家携手组建了"中国肿瘤基因组研究协作组(CCGC)",启动了中国肿瘤基因组研究项目并选择我国常见高发肿瘤胃癌作为参与ICGC的研究任务,拟采用外显子测序和典型病例的全基因组测序的方案.
在过去的3年中,运用外显子测序的研究方法,研究人员在卡波西肉瘤、小细胞肺癌、非小细胞肺癌、Hodgkin淋巴瘤、急性髓性白血病、小叶基底乳癌、慢性淋巴细胞性白血病、骨癌、葡萄膜黑色素瘤等肿瘤的研究中取得了显著的成就[42~48].
目前我国在白血病、黑色素瘤、多发性内分泌腺瘤综合征/家族性甲状腺髓样癌的研究中也取得了一定的成绩[43,49,50].
3.
3.
2运用新一代测序技术研究肿瘤的案例新一代测序技术可以用于研究肿瘤DNA,以发现核苷酸序列的改变和染色体结构改变,例如Harbour等[48]利用NimbleGen2.
1M芯片捕获外显子,GenomeAnalyzerII测序系统对31例转移性葡萄膜黑色素瘤患者测序,在84%的转移性肿瘤中发现了BAP1基因的失活性体细胞突变.
肿瘤DNA序列存在染色体易位、倒位、重复和缺失,随着双向测序的出现,可以通过组装,确定DNA片段的物理距离的原理研究肿瘤DNA的结构变异,Stephen等[47]研究发现许多肿瘤中2%~3%的患者存在大量的基因组重排现象.
同时可以通过对原发灶、转移灶和异种移植物DNA测序,研究肿瘤的进展情况,例如对基底样乳腺癌的原发肿瘤和转移灶或异种移植物测序发现,在转移灶或异种移植物的突变来源于原发灶,但是会选择性扩增[51].
而小叶乳腺癌的转移灶的多数突变(约60%)不存在原发灶中[44].
4在基因诊断中的运用由于全基因组外显子测序技术在孟德尔疾病和复杂疾病研究中的优势,目前已有成功运用该技术进行基因诊断和分子诊断的报道.
Choi等[52]通过NimbleGen2.
1M芯片和Illumina测序系统,对疑似Bartter综合征的5例高加索人进行测序,结果发现其中1例患者的SLC26A3基因(为先天性氯腹泻的位点)的一个错义突变D652N,说明该患者是先天性氯腹泻,而不是Bartter综合征,其临床也支持这个诊断.
后期对39例疑似Bartter综合征和95例对照进行Sanger测序验证,发现其中5例患者是SLC26A3纯合子突变,且临床表现也符合先天性氯腹泻诊断.
上述研究表明全基因组外显子测序可以用于发现疾病相关的基因信息,利用测序信息结合临床特征可对相关遗传病做出正确的诊断,从而进一步证852HEREDITAS(Beijing)2011第33卷明全基因组外显子测序能运用于疾病的分子诊断.
除此之外,还有利用全基因组外显子测序的策略对其他临床上难诊断的疾病进行诊断的相关报道,如新生儿糖尿病、难治性炎性肠病和Charcot-Marie-Toothatrophy综合征的分子水平的诊断[53-55].
5展望目前,全基因组外显子测序已在孟德尔疾病或罕见综合征的研究中取得了重大突破,证实了全基因组外显子测序对鉴定孟德尔疾病或罕见综合征的致病基因是行之有效的;可以运用在复杂疾病的研究中,目前在肿瘤方面的研究较多,但由于该技术存在一些尚待完善的问题,导致全基因组外显子测序在常见疾病的研究进展缓慢,但随着技术的发展和后续分析手段的丰富,预期在不久的将来,外显子测序会广泛地运用到其他复杂疾病的研究中;也可以用于疾病的分子诊断,为临床一些难以诊断的疾病提供了一种新诊断思路.
因此,Biesecker等[56]在NatureGenetics发表评论说:外显子测序使得医学基因组学成为现实.
全基因组外显子测序也存在不足之处:(1)该技术对结构变异与非编码区变异的研究具有局限性,而结构变异和非编码区变异也可能与疾病相关,可通过全基因组测序研究非编码区变异,同时进行基因组组装,同基因序列对比后发现一些结构变异,如CNV、indels,此外还可以结合芯片研究检测CNV;(2)在目标区域的捕获时,存在捕获不均、捕获偏差等现象,可以通过增加测序深度,获得更多的序列信息进行统计分析,以尽可能的弥补这些偏差;(3)研究常见疾病的罕见突变时,需要庞大的样本量,也导致了测序的费用的升高;目前用于验证的基因分型的平台如Sequenom、TaqMan主要适用于研究常见变异,需要定制芯片或通过Sanger测序,但这些方法耗时和价格较昂贵;数据分析的方法仍然不够完善,尚待解决,难以从海量的数据中迅速发现具有重大价值的信息.
尽管如此,由于外显子是与疾病及表型相关的最具特征性的区域,并且迄今为止较难评价非编码区域对疾病的影响,所以在全基因组测序费用居高不下的今天,全基因组外显子测序仍然不失为一个很好的选择.
外显子测序为阐明疾病的发病机理提供了新的线索,在疾病的基因诊断和致病基因的研究方面有广阔的前景,为之后的功能研究奠定了理论基础,从而为临床应用铺平了道路.
参考文献(References):[1]KuCS,LoyEY,PawitanY,ChiaKS.
Thepursuitofge-nome-wideassociationstudies:wherearewenowJHumGenet,2010,55(4):195–206.
[2]McClellanJM,SusserE,KingMC.
Schizophrenia:acommondiseasecausedbymultiplerarealleles.
BrJPsychiatry,2007,190:194–199.
[3]McClellanJ,KingMC.
Geneticheterogeneityinhumandisease.
Cell,2010,141(2):210–217.
[4]BotsteinD,RischN.
Discoveringgenotypesunderlyinghumanphenotypes:pastsuccessesformendeliandisease,futureapproachesforcomplexdisease.
NatGenet,2003,33(3):228–237.
[5]StensonPD,BallEV,MortM,PhillipsAD,ShielJA,ThomasNST,AbeysingheS,KrawczakM,CooperDN.
Humangenemutationdatabase(HGMD):2003update.
HumMutat,2003,21(6):577–581.
[6]KahvejianA,QuackenbushJ,ThompsonJF.
WhatwouldyoudoifyoucouldsequenceeverythingNatBiotechnol,2008,26(10):1125–1133.
[7]GnirkeA,MelnikovA,MaguireJ,RogovP,LeProustEM,BrockmanW,FennellT,GiannoukosG,FisherS,RussC,GabrielS,JaffeDB,LanderES,NusbaumC.
Solutionhybridselectionwithultra–longoligonucleotidesformas-sivelyparalleltargetedsequencing.
NatBiotechnol,2009,27(2):182–189.
[8]ShendureJ,JiH.
Next–generationDNAsequencing.
NatBiotechnol,2008,26(10):1135–1145.
[9]HillierLW,MarthGT,QuinlanAR,DoolingD,FewellG,BarnettD,FoxP,GlasscockJI,HickenbothamM,HuangW,MagriniVJ,RichtRJ,SanderSN,StewartDA,Strom-bergM,TsungEF,WylieT,SchedlT,WilsonRK,MardisER.
Whole–genomesequencingandvariantdiscoveryinC.
elegans.
NatMethods,2008,5(2):183–188.
[10]DroegeM,HillB.
TheGenomeSequencerFLXSys-tem–longerreads,moreapplications,straightforwardbioinformaticsandmorecompletedatasets.
JBiotechnol,2008,136(1–2):3–10.
[11]HashimotoS,QuW,AhsanB,OgoshiK,SasakiA,Naka-taniY,LeeY,OgawaM,AmetaniA,SuzukiY,SuganoS,LeeCC,NutterRC,MorishitaS,MatsushimaK.
High–resolutionanalysisofthe5'–endtranscriptomeusinganextgenerationDNAsequencer.
PLoSOne,2009,4(1):e4108.
第8期张鑫等:全基因组外显子测序及其应用853[12]RothbergJM,LeamonJH.
Thedevelopmentandimpactof454sequencing.
NatBiotechnol,2008,26(10):1117–1124.
[13]NgPC,LevyS,HuangJQ,StockwellTB,WalenzBP,LiK,AxelrodN,BusamDA,StrausbergRL,VenterJC.
Ge-neticvariationinanindividualhumanexome.
PLoSGenet,2008,4(8):e1000160.
[14]LiYR,VinckenboschN,TianG,Huerta–SanchezE,JiangT,JiangH,AlbrechtsenA,AndersenG,CaoHZ,KorneliussenT,GrarupN,GuoYR,HellmanI,JinX,LiQB,LiuJT,LiuX,SparsoT,TangMF,WuHL,WuRH,YuC,ZhengHC,As-trupA,BolundL,HolmkvistJ,JrgensenT,KristiansenK,SchmitzO,SchwartzTW,ZhangXQ,LiRQ,YangHM,WangJ,HansenT,PedersenO,NielsenR,WangJ.
Rese-quencingof200humanexomesidentifiesanexcessoflow–frequencynon–synonymouscodingvariants.
NatGenet,2010,42(11):969–972.
[15]NgSB,TurnerEH,RobertsonPD,FlygareSD,BighamAW,LeeC,ShafferT,WongM,BhattacharjeeA,EichlerEE,BamshadM,NickersonDA,ShendureJ.
Targetedcaptureandmassivelyparallelsequencingof12humanexomes.
Na-ture,2009,461(7261):272–276.
[16]WangJL,YangX,XiaK,HuZM,WengL,JinX,JiangH,ZhangP,ShenL,GuoJF,LiN,LiYR,LeiLF,ZhouJ,DuJ,ZhouYF,PanQ,WangJ,WangJ,LiRQ,TangBS.
TGM6identifiedasanovelcausativegeneofspinocerebellarataxiasusingexomesequencing.
Brain,2010,133(Pt12):3510–3518.
[17]LiuY,GaoM,LvYM,YangX,RenYQ,JiangT,ZhangX,GuoBR,LiM,ZhangQ,ZhangP,ZhouFS,ChenG,YinXY,ZuoXB,SunLD,ZhengXD,ZhangSM,LiuJJ,ZhouYW,LiYR,WangJ,YangHM,YangS,LiRQ,ZhangXJ.
Con-firmationbyExomeSequencingofthePathogenicRoleofNCSTNMutationsinAcneInversa(HidradenitisSuppura-tiva).
JInvestDermatol,2011,131(7):1570–1572.
[18]NgSB,BighamAW,BuckinghamKJ,HannibalMC,McMil-linMJ,GildersleeveHI,BeckAE,TaborHK,CooperGM,MeffordHC,LeeC,TurnerEH,SmithJD,RiederMJ,Yo-shiuraKI,MatsumotoN,OhtaT,NiikawaN,NickersonDA,BamshadMJ,ShendureJ.
ExomesequencingidentifiesMLL2mutationsasacauseofKabukisyndrome.
NatGenet,2010,42(9):790–793.
[19]HoischenA,vanBonBW,GilissenC,ArtsP,vanLierB,SteehouwerM,deVriesP,deReuverR,WieskampN,Mor-tierG,DevriendtK,AmorimMZ,RevencuN,KiddA,Bar-bosaM,TurnerA,SmithJ,OleyC,HendersonA,HayesIM,ThompsonEM,BrunnerHG,deVriesBBA,VeltmanJA.
DenovomutationsofSETBP1causeSchinzel–Giedionsyn-drome.
NatGenet,2010,42(6):483–485.
[20]GilissenC,ArtsHH,HoischenA,SpruijtL,MansDA,ArtsP,vanLierB,SteehouwerM,vanReeuwijkJ,KantSG,Roep-manR,KnoersNVAM,VeltmanJA,BrunnerHG.
ExomesequencingidentifiesWDR35variantsinvolvedinSensen-brennersyndrome.
AmJHumGenet,2010,87(3):418–423.
[21]LalondeE,AlbrechtS,HaKCH,JacobK,BolducN,Poly-chronakosC,DechelotteP,MajewskiJ,JabadoN.
Unex-pectedallelicheterogeneityandspectrumofmutationsinFowlersyndromerevealedbynext–generationexomese-quencing.
HumMutat,2010,31(8):918–923.
[22]PierceSB,WalshT,ChisholmKM,LeeMK,ThorntonAM,FiumaraA,OpitzJM,Levy–LahadE,KlevitRE,KingMC.
MutationsintheDBP–deficiencyproteinHSD17B4causeovariandysgenesis,hearingloss,andataxiaofPerraultSyn-drome.
AmJHumGenet,2010,87(2):282–288.
[23]SimpsonMA,IrvingMD,AsilmazE,GrayMJ,DafouD,ElmslieFV,MansourS,HolderSE,BrainCE,BurtonBK,KimKH,PauliRM,AftimosS,StewartH,KimCA,Holder–EspinasseM,RobertsonSP,DrakeWM,TrembathRC.
MutationsinNOTCH2causeHajdu–Cheneysyndrome,adisorderofsevereandprogressiveboneloss.
NatGenet,2011,43(4):303–305.
[24]BeckerJ,SemlerO,GilissenC,LiY,BolzHJ,GiuntaC,BergmannC,RohrbachM,KoerberF,ZimmermannK,deVriesP,WirthB,SchoenauE,WollnikB,VeltmanJA,HoischenA,NetzerC.
Exomesequencingidentifiestruncat-ingmutationsinHumanSERPINF1inAutosomal–Recessiveosteogenesisimperfecta.
AmJHumGenet,2011,88(3):362–371.
[25]HaackTB,DanhauserK,HaberbergerB,HoserJ,StreckerV,BoehmD,UzielG,LamanteaE,InvernizziF,PoultonJ,RolinskiB,IusoA,BiskupS,SchmidtT,MewesHW,WittigI,MeitingerT,ZevianiM,ProkischH.
ExomesequencingidentifiesACAD9mutationsasacauseofcomplexIdefi-ciency.
NatGenet,2010,42(12):1131–1134.
[26]NgSB,BuckinghamKJ,LeeC,BighamAW,TaborHK,DentKM,HuffCD,ShannonPT,JabsEW,NickersonDA,Shen-dureJ,BamshadMJ.
Exomesequencingidentifiesthecauseofamendeliandisorder.
NatGenet,2009,42(1):30–35.
[27]JohnsonJO,GibbsJR,vanMaldergemL,HouldenH,Sin-gletonAB.
Exomesequencinginbrown–vialetto–vanlaeresyndrome.
AmJHumGenet,2010,87(4):567–569.
[28]KrawitzPM,SchweigerMR,RdelspergerC,MarcelisC,KlschU,MeiselC,StephaniF,KinoshitaT,MurakamiY,BauerS,IsauM,FischerA,DahlA,KerickM,HechtJ,KhlerS,JgerM,GrünhagenJ,deCondorBJ,DoelkenS,854HEREDITAS(Beijing)2011第33卷BrunnerHG,MeineckeP,PassargeE,ThompsonMD,ColeDE,HornD,RoscioliT,MundlosS,RobinsonPN.
Iden-tity–by–descentfilteringofexomesequencedataidentifiesPIGVmutationsinhyperphosphatasiamentalretardationsyndrome.
NatGenet,2010,42(10):827–829.
[29]MusunuruK,PirruccelloJP,DoR,PelosoGM,GuiducciC,SougnezC,GarimellaKV,FisherS,AbreuJ,BarryAJ,FennellT,BanksE,AmbrogioL,CibulskisK,KernytskyA,GonzalezE,RudziczN,EngertJC,DepristoMA,DalyMJ,CohenJC,HobbsHH,AltshulerD,SchonfeldG,GabrielSB,YueP,KathiresanS.
Exomesequencing,ANGPTL3muta-tions,andfamilialcombinedHypolipidemia.
NEnglJMed,2010,363(23):2220–2227.
[30]ZüchnerS,DallmanJ,WenR,BeechamG,NajA,FarooqA,KohliMA,WhiteheadPL,HulmeW,KonidariI,EdwardsYJK,CaiGQ,PeterI,SeoD,BuxbaumJD,HainesJL,BlantonS,YoungJ,AlfonsoE,VanceJM,LamBL,Peri-cak–VanceMA.
Whole–ExomesequencinglinksavariantinDHDDStoretinitispigmentosa.
AmJHumGenet,2011,88(2):201–206.
[31]WalshT,ShahinH,Elkan–MillerT,LeeMK,ThorntonAM,RoebW,AbuRayyanA,LoulusS,AvrahamKB,KingMC,KanaanM.
WholeexomesequencingandhomozygositymappingidentifymutationinthecellpolarityproteinGPSM2asthecauseofnonsyndromichearinglossDFNB82.
AmJHumGenet,2010,87(1):90–94.
[32]OttoEA,HurdTW,AirikR,ChakiM,ZhouWB,StoetzelC,PatilSB,LevyS,GhoshAK,Murga–ZamalloaCA,vanReeuwijkJ,LetteboerSJF,SangLY,GilesRH,LiuQ,CoeneKLM,Estrada–CuzcanoA,CollinRWJ,McLaughlinHM,HeldS,KasanukiJM,RamaswamiG,ConteJ,LopezI,WashburnJ,MacDonaldJ,HuJH,YamashitaY,MaherER,Guay–WoodfordLM,NeumannHPH,ObermüllerN,KoenekoopRK,BergmannC,BeiXS,LewisRA,KatsanisN,LopesV,WilliamsDS,LyonsRH,DangCV,BritoDA,DiasMB,ZhangXM,CavalcoliJD,NürnbergG,NürnbergP,PierceEA,JacksonPK,AntignacC,SaunierS,RoepmanR,DollfusH,KhannaH,HildebrandtF.
Candidateexomecap-tureidentifiesmutationofSDCCAG8asthecauseofareti-nal–renalciliopathy.
NatGenet,2010,42(10):840–850.
[33]SirmaciA,WalshT,AkayH,SpiliopoulosM,akalarYB,Hasanefendiolu–BayrakA,DumanD,FarooqA,KingMC,TekinM.
MASP1mutationsinpatientswithfacial,umbilical,coccygeal,andauditoryfindingsofcarnevale,malpuech,OSA,andmichelssyndromes.
AmJHumGenet,2010,87(5):679–686.
[34]OstergaardP,SimpsonMA,BriceG,MansourS,ConnellFC,OnoufriadisA,ChildAH,HwangJ,KalidasK,MortimerPS,TrembathR,JefferyS.
RapididentificationofmutationsinGJC2inprimarylymphoedemausingwholeexomesequenc-ingcombinedwithlinkageanalysiswithdelineationofthephenotype.
JMedGenet,2011,48(4):251–255.
[35]JohnsonJO,MandrioliJ,BenatarM,AbramzonY,vanDeer-linVM,TrojanowskiJQ,GibbsJR,BrunettiM,GronkaS,WuuJ,DingJH,McCluskeyL,Martinez–LageM,FalconeD,HernandezDG,ArepalliS,ChongS,SchymickJC,RothsteinJ,LandiF,WangYD,CalvoA,MoraG,SabatelliM,Mon-surròMR,BattistiniS,SalviF,SpataroR,SolaP,BorgheroG,GalassiG,ScholzSW,TaylorJP,RestagnoG,ChioA,TraynorBJ.
ExomesequencingrevealsVCPmutationsasacauseoffamilialALS.
Neuron,2010,68(5):857–864.
[36]CaliskanM,ChongJX,UricchioL,AndersonR,ChenP,SougnezC,GarimellaK,GabrielSB,dePristoMA,ShakirK,MaternD,DasS,WaggonerD,NicolaeDL,OberC.
Exomesequencingrevealsanovelmutationforautosomalrecessivenon–syndromicmentalretardationintheTECRgeneonchromosome19p13.
HumMolGenet,2010,20(7):1285–1289.
[37]AnastasioN,Ben–OmranT,TeebiA,HaKCH,LalondeE,AliR,AlmureikhiM,DerKaloustianVM,LiuJH,RosenblattDS,MajewskiJ,Jerome–MajewskaLA.
MutationsinSCARF2areresponsibleforVanDenEnde–Guptasyndrome.
AmJHumGenet,2010,87(4):553–559.
[38]BolzeA,ByunM,McDonaldD,MorganNV,AbhyankarA,PremkumarL,PuelA,BaconCM,Rieux–LaucatF,PangK,BritlandA,AbelL,CantA,MaherER,RiedlSJ,HambletonS,CasanovaJL.
Whole-Exome-Sequencing-BaseddiscoveryofHumanFADDdeficiency.
AmJHumGenet,2010,87(6):873–881.
[39]BowdenDW,AnSS,PalmerND,BrownWM,NorrisJM,HaffnerSM,HawkinsGA,GuoX,RotterJI,ChenYD,WagenknechtLE,LangefeldCD.
Molecularbasisofalink-agepeak:exomesequencingandfamily–basedanalysisiden-tifyararegeneticvariantintheADIPOQgeneintheIRASFamilyStudy.
HumMolGenet,201019(20):4112–4120.
[40]BilgüvarK,zturkAK,LouviA,KwanKY,ChoiM,TatliB,YalnizoluD,TüysüzB,alayanAO,GkbenS,Kay-makalanH,BarakT,BakircioluM,YasunoK,HoW,SandersS,ZhuY,YilmazS,DinerA,JohnsonMH,BronenRA,KoerN,PerH,ManeS,PamirMN,YalinkayaC,Ku-mandaS,TopuM,zmenM,estanN,LiftonRP,StateMW,GünelM.
Whole–exomesequencingidentifiesrecessiveWDR62mutationsinseverebrainmalformations.
Nature,2010,467(7312):207–210.
第8期张鑫等:全基因组外显子测序及其应用855[41]HudsonTJ,AndersonW,ArtezA,BarkerAD,BellC,Bern-abéRR,BhanMK,CalvoF,EerolaI,GerhardDS,Gutt-macherA,GuyerM,HemsleyFM,JenningsJL,KerrD,KlattP,KolarP,KusadaJ,LaneDP,LaplaceF,YouyongL,Net-tekovenG,OzenbergerB,PetersonJ,RaoTS,RemacleJ,SchaferAJ,ShibataT,StrattonMR,VockleyJG,WatanabeK,YangH,YuenMM,KnoppersBM,BobrowM,Cam-bon–ThomsenA,DresslerLG,DykeSO,JolyY,KatoK,KennedyKL,NicolásP,ParkerMJ,Rial–SebbagE,Ro-meo–CasabonaCM,ShawKM,WallaceS,WiesnerGL,ZepsN,LichterP,BiankinAV,ChabannonC,ChinL,ClémentB,deAlavaE,DegosF,FergusonML,GearyP,HayesDN,HudsonTJ,JohnsAL,KasprzykA,NakagawaH,PennyR,PirisMA,SarinR,ScarpaA,vandeVijverM,FutrealPA,AburataniH,BayésM,BotwellDD,CampbellPJ,EstivillX,GerhardDS,GrimmondSM,GutI,HirstM,López–OtínC,MajumderP,MarraM,McPhersonJD,NakagawaH,NingZ,PuenteXS,RuanY,ShibataT,StrattonMR,StunnenbergHG,SwerdlowH,VelculescuVE,WilsonRK,XueHH,YangL,SpellmanPT,BaderGD,BoutrosPC,CampbellPJ,FlicekP,GetzG,GuigóR,GuoG,HausslerD,HeathS,HubbardTJ,JiangT,JonesSM,LiQ,López–BigasN,LuoR,Mut-huswamyL,OuelletteBF,PearsonJV,PuenteXS,QuesadaV,RaphaelBJ,SanderC,ShibataT,SpeedTP,SteinLD,StuartJM,TeagueJW,TotokiY,TsunodaT,ValenciaA,WheelerDA,WuH,ZhaoS,ZhouG,SteinLD,GuigóR,HubbardTJ,JolyY,JonesSM,KasprzykA,LathropM,López–BigasN,OuelletteBF,SpellmanPT,TeagueJW,ThomasG,ValenciaA,YoshidaT,KennedyKL,AxtonM,DykeSO,FutrealPA,GerhardDS,GunterC,GuyerM,HudsonTJ,McPhersonJD,MillerLJ,OzenbergerB,ShawKM,KasprzykA,SteinLD,ZhangJ,HaiderSA,WangJ,YungCK,CrossA,LiangY,GnaneshanS,GubermanJ,HsuJ,BobrowM,ChalmersDR,HaselKW,JolyY,KaanTS,KennedyKL,KnoppersBM,LowranceWW,MasuiT,NicolásP,Rial–SebbagE,Rodri-guezLL,VergelyC,YoshidaT,GrimmondSM,BiankinAV,BowtellDD,CloonanN,deFazioA,EshlemanJR,Etemad-moghadamD,GardinerBA,KenchJG,ScarpaA,SutherlandRL,TemperoMA,WaddellNJ,WilsonPJ,McPhersonJD,GallingerS,TsaoMS,ShawPA,PetersenGM,Muk-hopadhyayD,DePinhoRA,ThayerS,ShazandK,BeckT,SamM,TimmsL,BallinV,LuY,JiJ,ZhangX,ChenF,HuX,ZhouG,YangQ,TianG,ZhangL,XingX,LiX,ZhuZ,YuY,YuJ,YangH,LathropM,TostJ,BrennanP,HolcatovaI,ZaridzeD,BrazmaA,EgevardL,ProkhortchoukE,BanksRE,UhlénM,Cambon–ThomsenA,ViksnaJ,PontenF,SkryabinK,StrattonMR,FutrealPA,BirneyE,BorgA,Borresen–DaleAL,CaldasC,FoekensJA,MartinS,Reis–FilhoJS,RichardsonAL,SotiriouC,StunnenbergHG,ThomsG,vandeVijverM,van'tVeerL,CalvoF,BirnbaumD,BlancheH,BoucherP,BoyaultS,ChabannonC,GutI,Masson–JacquemierJD,LathropM,PauportéI,PivotX,Vincent–SalomonA,TaboneE,TheilletC,ThomasG,TostJ,TreilleuxI,Bioulac–SageP,ClémentB,DecaensT,DegosF,FrancoD,GutI,GutM,HeathS,LathropM,SamuelD,ThomasG,Zucman–RossiJ,LichterP,EilsR,BrorsB,KorbelJO,KorshunovA,LandgrafP,LehrachH,PfisterS,RadlwimmerB,ReifenbergerG,TaylorMD,vonKalleC,MajumderPP,SarinR,RaoTS,BhanMK,ScarpaA,Peder-zoliP,LawlorRA,DelledonneM,BardelliA,BiankinAV,GrimmondSM,GressT,KlimstraD,ZamboniG,ShibataT,NakamuraY,NakagawaH,KusadaJ,TsunodaT,MiyanoS,AburataniH,KatoK,FujimotoA,YoshidaT,CampoE,López–OtínC,EstivillX,GuigóR,deSanjoséS,PirisMA,MontserratE,González–DíazM,PuenteXS,JaresP,Valen-ciaA,HimmelbaueH,QuesadaV,BeaS,StrattonMR,Fut-realPA,CampbellPJ,Vincent–SalomonA,RichardsonAL,Reis–FilhoJS,vandeVijverM,ThomasG,Mas-son–JacquemierJD,AparicioS,BorgA,Brresen–DaleAL,CaldasC,FoekensJA,StunnenbergHG,van'tVeerL,EastonDF,SpellmanPT,MartinS,BarkerAD,ChinL,CollinsFS,ComptonCC,FergusonML,GerhardDS,GetzG,GunterC,GuttmacherA,GuyerM,HayesDN,LanderES,OzenbergerB,PennyR,PetersonJ,SanderC,ShawKM,SpeedTP,SpellmanPT,VockleyJG,WheelerDA,WilsonRK,HudsonTJ,ChinL,KnoppersBM,LanderES,LichterP,SteinLD,StrattonMR,AndersonW,BarkerAD,BellC,BobrowM,BurkeW,CollinsFS,ComptonCC,DePinhoRA,EastonDF,FutrealPA,GerhardDS,GreenAR,GuyerM,HamiltonSR,HubbardTJ,KallioniemiOP,KennedyKL,LeyTJ,LiuET,LuY,MajumderP,MarraM,OzenbergerB,PetersonJ,SchaferAJ,SpellmanPT,StunnenbergHG,WainwrightBJWilsonRK,YangH.
Internationalnetworkofcancergenomeprojects.
Nature,2010,464(7291):993–998.
[42]ByunM,AbhyankarA,LelargeV,PlancoulaineS,PalanduzA,TelhanL,BoissonB,PicardC,DewellS,ZhaoCN,JouanguyE,FeskeS,AbelL,CasanovaJL.
Whole–exomesequencing–baseddiscoveryofSTIM1deficiencyinachildwithfatalclassicKaposisarcoma.
JExpMed,2010,207(11):2307–2312.
[43]YanXJ,XuJ,GuZH,PanCM,LuG,ShenY,ShiJY,ZhuYM,TangL,ZhangXW,LiangWX,MiJQ,SongHD,LiKQ,ChenZ,ChenSJ.
ExomesequencingidentifiessomaticmutationsofDNAmethyltransferasegeneDNMT3Ainacute856HEREDITAS(Beijing)2011第33卷monocyticleukemia.
NatGenet,2011,43(4):309–315.
[44]ShahSP,MorinRD,KhattraJ,PrenticeL,PughT,BurleighA,DelaneyA,GelmonK,GulianyR,SenzJ,SteidlC,HoltRA,JonesS,SunM,LeungG,MooreR,SeversonT,TaylorGA,TeschendorffAE,TseK,TurashviliG,VarholR,WarrenRL,WatsonP,ZhaoYJ,CaldasC,HuntsmanD,HirstM,MarraMA,AparicioS.
Mutationalevolutioninalobularbreasttumourprofiledatsinglenucleotideresolution.
Nature,2009,461(7265):809–813.
[45]LeeW,JiangZS,LiuJF,HavertyPM,GuanYH,StinsonJ,YueP,ZhangY,PantKP,BhattD,HaC,JohnsonS,Kenne-merMI,MohanS,NazarenkoI,WatanabeC,SparksAB,ShamesDS,GentlemanR,deSauvageFJ,SternH,PanditaA,BallingerDG,DrmanacR,ModrusanZ,SeshagiriS,ZhangZM.
Themutationspectrumrevealedbypairedgenomese-quencesfromalungcancerpatient.
Nature,2010,465(7297):473–477.
[46]SaarinenS,AavikkoM,AittomkiK,LaunonenV,LehtonenR,FranssilaK,LehtonenHJ,KaasinenE,BroderickP,Tark-kanenJ,BainBJ,BauduerF,UnalA,SwerdlowAJ,CookeR,MakinenMJ,HoulstonR,VahteristoP,AaltonenLA.
ExomesequencingrevealsgermlineNPATmutationasacandidateriskfactorforHodgkinlymphoma.
Blood,2011.
[47]StephensPJ,GreenmanCD,FuBY,YangFT,BignellGR,MudieLJ,PleasanceED,LauKW,BeareD,StebbingsLA,McLarenS,LinML,McBrideDJ,VarelaI,Nik–ZainalS,LeroyC,JiaMM,MenziesA,ButlerAP,TeagueJW,QuailMA,BurtonJ,SwerdlowH,CarterNP,MorsbergerLA,Iacobuzio–DonahueC,FollowsGA,GreenAR,FlanaganAM,StrattonMR,FutrealPA,CampbellPJ.
Massivege-nomicrearrangementacquiredinasinglecatastrophiceventduringcancerdevelopment.
Cell,2011,144(1):27–40.
[48]HarbourJW,OnkenMD,RobersonEDO,DuanSH,CaoL,WorleyLA,CouncilML,MatatallKA,HelmsC,BowcockAM.
FrequentmutationofBAP1inmetastasizinguvealmelanomas.
Science,2010,330(6009):1410–1413.
[49]WeiXM,WaliaV,LinJC,TeerJK,PrickettTD,GartnerJ,DavisS,Stemke–HaleK,DaviesMA,GershenwaldJE,RobinsonW,RobinsonS,RosenbergSA,SamuelsY.
ExomesequencingidentifiesGRIN2Aasfrequentlymutatedinmelanoma.
NatGenet,2011,43(5):442–446.
[50]QiXP,MaJM,DuZF,YingRB,FeiJ,JinHY,HanJS,WangJQ,ChenXL,ChenCY,LiuWT,LuJJ,ZhangJG,ZhangXN.
RETgermlinemutationsidentifiedbyexomesequencinginaChinesemultipleendocrineneoplasiaType2A/Familialme-dullarythyroidcarcinomafamily.
PLoSOne,2011,6(5):e20353.
[51]DingL,EllisMJ,LiSQ,LarsonDE,ChenK,WallisJW,HarrisCC,McLellanMD,FultonRS,FultonLL,AbbottRM,HoogJ,DoolingDJ,KoboldtDC,SchmidtH,KalickiJ,ZhangQY,ChenL,LinL,WendlMC,McMichaelJF,Ma-griniVJ,CookL,McGrathSD,VickeryTL,AppelbaumE,DeschryverK,DaviesS,GuintoliT,LinL,CrowderR,TaoY,SniderJE,SmithSM,DukesAF,SandersonGE,PohlCS,DelehauntyKD,FronickCC,PapeKA,ReedJS,RobinsonJS,HodgesJS,SchierdingW,DeesND,ShenD,LockeDP,WiechertME,EldredJM,PeckJB,OberkfellBJ,LolofieJT,DuFY,HawkinsAE,O'LaughlinMD,BernardKE,Cun-ninghamM,ElliottG,MasonMD,ThompsonDMJr,Ivano-vichJL,GoodfellowPJ,PerouCM,WeinstockGM,AftR,WatsonM,LeyTJ,WilsonRK,MardisER.
Genomeremod-ellinginabasal–likebreastcancermetastasisandxenograft.
Nature,2010,464(7291):999–1005.
[52]ChoiM,SchollUI,JiWZ,LiuTW,TikhonovaIR,ZumboP,NayirA,BakkaloluA,zenS,SanjadS,Nelson–WilliamsC,FarhiA,ManeS,LiftonRP.
GeneticdiagnosisbywholeexomecaptureandmassivelyparallelDNAsequencing.
ProcNatlAcadSciUSA,2009,106(45):19096–19101.
[53]WortheyEA,MayerAN,SyversonGD,HelblingD,BonacciBB,DeckerB,SerpeJM,DasuT,TschannenMR,VeithRL,BasehoreMJ,BroeckelU,Tomita–MitchellA,ArcaMJ,CasperJT,MargolisDA,BickDP,HessnerMJ,RoutesJM,VerbskyJW,JacobHJ,DimmockDP.
Makingadefinitivediagnosis:Successfulclinicalapplicationofwholeexomesequencinginachildwithintractableinflammatoryboweldisease.
GenetMed,2010,13(3):255–262.
[54]BonnefondA,DurandE,SandO,DeGraeveF,GallinaS,BusiahK,LobbensS,SimonA,Bellanné–ChantelotC,LétourneauL,ScharfmannR,DelplanqueJ,SladekR,PolakM,VaxillaireM,FroguelP.
Moleculardiagnosisofneonataldiabetesmellitususingnext–generationsequencingofthewholeexome.
PLoSOne,2010,5(10):e13630.
[55]MontenegroG,PowellE,HuangJ,SpezianiF,EdwardsYJK,BeechamG,HulmeW,SiskindC,VanceJ,ShyM,ZüchnerS.
ExomesequencingallowsforrapidgeneidentificationinaCharcot–Marie–Toothfamily.
AnnNeurol,2011,69(3):464–470.
[56]BieseckerLG.
Exomesequencingmakesmedicalgenomicsareality.
NatGenet,42(1):13–14.
无忧云-河南洛阳BGP,CEPH集群分布式存储,数据安全可靠,活动期间月付大优惠!
无忧云怎么样?无忧云服务器好不好?无忧云值不值得购买?无忧云是一家成立于2017年的老牌商家旗下的服务器销售品牌,现由深圳市云上无忧网络科技有限公司运营,是正规持证IDC/ISP/IRCS商家,主要销售国内、中国香港、国外服务器产品,线路有腾讯云国外线路、自营香港CN2线路等,都是中国大陆直连线路,非常适合免备案建站业务需求和各种负载较高的项目,同时国内服务器也有多个BGP以及高防节点...

DiyVM(50元起)老牌商家,香港沙田CN2直连vps/不限流量/五折终身优惠
diyvm怎么样?diyvm是一家国内成立时间比较久的主机商家了,大约在6年前站长曾经用过他家的美国机房的套餐,非常稳定,适合做站,目前商家正在针对香港沙田机房的VPS进行促销,给的是五折优惠,续费同价,香港沙田机房走的是CN2直连的线路,到大陆地区的速度非常好,DiyVM商家采用小带宽不限流量的形式,带宽2Mbps起步,做站完全够用,有需要的朋友可以入手。diyvm优惠码:五折优惠码:OFF50...
BuyVM新设立的迈阿密机房速度怎么样?简单的测评速度性能
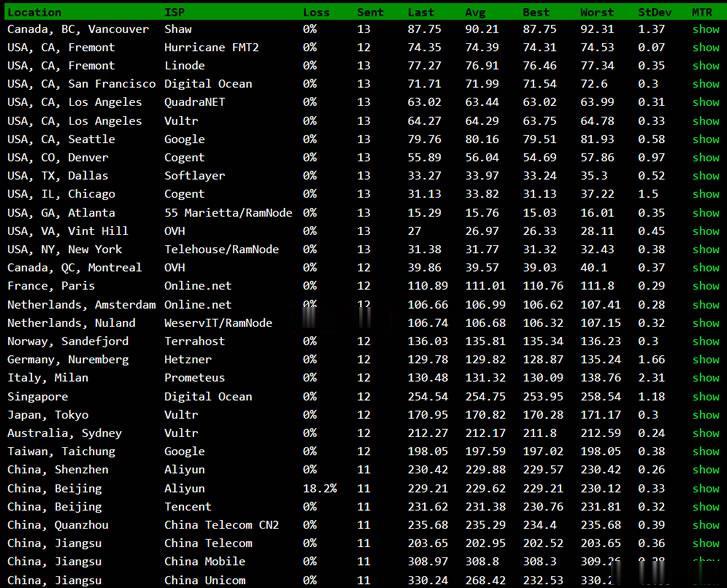
BuyVM商家算是一家比较老牌的海外主机商,公司设立在加拿大,曾经是低价便宜VPS主机的代表,目前为止有提供纽约、拉斯维加斯、卢森堡机房,以及新增加的美国迈阿密机房。如果我们有需要选择BuyVM商家的机器需要注意的是注册信息的时候一定要规范,否则很容易出现欺诈订单,甚至你开通后都有可能被禁止账户,也是这个原因,曾经被很多人吐槽的。这里我们简单的对于BuyVM商家新增加的迈阿密机房进行简单的测评。如...
jpboy为你推荐
-
iphone6s内存多大iphone6s plus内存多大可爱桌面背景图片求好看的桌面背景图片cf蜗牛外挂我想让cf用什么外挂可以让号被封了要最快那种。最好永久封了最好电脑管家和360哪个好电脑管家和360安全卫士哪个好帕萨特和迈腾哪个好2019帕萨特和迈腾哪个好?隔音怎么样?手机杀毒哪个好手机杀毒软件哪个好 五大手机安全软件杀毒大比拼网校哪个好初中网校哪个好?google广告申请怎么样申请谷歌广告成功google广告申请Google广告用户申请有何绝招?qq网盘在哪里QQ网盘在哪里